当前位置:
X-MOL 学术
›
Spectrochim. Acta. A Mol. Biomol. Spectrosc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
圆二色光谱评估在对比 pH 下蛋白质热展开后的结构变化:与分子动力学模拟的比较
Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy ( IF 4.3 ) Pub Date : 2022-02-23 , DOI: 10.1016/j.saa.2022.121039 Ponciano García-Gutiérrez 1 , Menandro Camarillo-Cadena 1 , Liliana I Vera-Robles 1 , Rafael A Zubillaga 1 , Andrés Hernández-Arana 1

"点击查看英文标题和摘要"
更新日期:2022-02-23
Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy ( IF 4.3 ) Pub Date : 2022-02-23 , DOI: 10.1016/j.saa.2022.121039 Ponciano García-Gutiérrez 1 , Menandro Camarillo-Cadena 1 , Liliana I Vera-Robles 1 , Rafael A Zubillaga 1 , Andrés Hernández-Arana 1
Affiliation
|
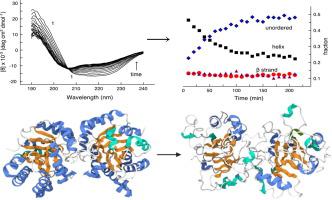
在大多数情况下,蛋白质展开事件的通常牢度阻碍了确定与此过程相关的二级结构的变化,因为这些确定依赖于高分辨率圆二色谱 (CD) 光谱的记录。在这项工作中,以 10 分钟间隔记录的远紫外 CD 光谱用于评估在不同 pH 条件下酵母丙糖磷酸异构酶 (yTIM) 的缓慢温度诱导展开中的四类二级结构的时间过程。CONTIN-LL 和 SELCON3 算法用于光谱的去卷积。两种算法都提供了根据一级动力学变化的螺旋和无序结构内容,与特定波长下 CD 数据显示的行为一致。未折叠 yTIM 光谱的分析,使用包含未折叠蛋白质光谱和两种算法之一的数据集,清楚地显示了在高 pH 值下更无序的蛋白质结构;对差异光谱的分析证实了这一发现。使用 AMBER 和 OPLS 力场进行的分子动力学 (MD) 模拟导致在高 pH 值下螺旋的更广泛损失和线圈的增益,这与光谱结果一致。然而,低 pH 值和高 pH 值展开的 yTIM 之间的结构差异相对较小。因此,CD 和 MD 的结果比较表明需要微调 MD 程序。使用 AMBER 和 OPLS 力场进行的分子动力学 (MD) 模拟导致在高 pH 值下螺旋的更广泛损失和线圈的增益,这与光谱结果一致。然而,低 pH 值和高 pH 值展开的 yTIM 之间的结构差异相对较小。因此,CD 和 MD 的结果比较表明需要微调 MD 程序。使用 AMBER 和 OPLS 力场进行的分子动力学 (MD) 模拟导致在高 pH 值下螺旋的更广泛损失和线圈的增益,这与光谱结果一致。然而,低 pH 值和高 pH 值展开的 yTIM 之间的结构差异相对较小。因此,CD 和 MD 的结果比较表明需要微调 MD 程序。

"点击查看英文标题和摘要"











































 京公网安备 11010802027423号
京公网安备 11010802027423号