当前位置:
X-MOL 学术
›
Chem. Bio. Drug Des.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
ETV4转录因子癌基因的生物信息学筛选和小分子抗癌药物的鉴定
Chemical Biology & Drug Design ( IF 3.2 ) Pub Date : 2021-11-10 , DOI: 10.1111/cbdd.13981 Ambily Nath I V 1 , Achuthsankar S Nair 1
Chemical Biology & Drug Design ( IF 3.2 ) Pub Date : 2021-11-10 , DOI: 10.1111/cbdd.13981 Ambily Nath I V 1 , Achuthsankar S Nair 1
Affiliation
|
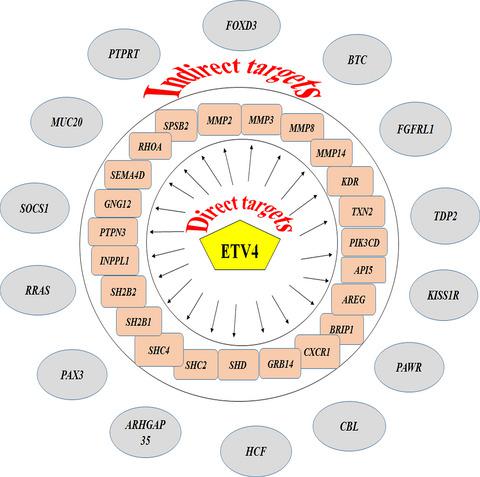
这项生物信息学研究旨在鉴定 ETV4 转录因子致癌基因并概述这些基因的抗癌药物。首先,我们收集了已知的 61 个 ETV4 癌症目标,这些目标被构建为两类查询,以筛选 GeneMANIA 中的多组学资源。该方法访问并在每组中添加了功能相似的 20 个基因。这些数据通过中枢基因、网络聚类、基因本体和通路分析进行解释,结果证实所有产生的基因都是癌症启动子。从这些基因的启动子区域鉴定出 ETS 结合基序。因此,计算出 23 个 ETV4 目标,并且将参与肿瘤发生的目标过滤为以下 16 个推定节点:MMP8、MMP14、KDR、BRIP1、CXCR1、GRB14、SHC2、SHC4、SH2B1、SH2B2、INPPL1、PTPN3、GNG12、SEMA4D、RHOA和SPSB2。这些癌基因的转录调控是由一个广泛的 miRNA 网络协调的,该网络发现可以解除对许多癌症通路的调控。使用 DgIb 数据库,高质量的 6 种癌基因药物组合(MMP8 -CHEMBL1231240、MMP8 -Aminomethylamide、CXCR1 -Reparixin、SEMA4D -Pepinemab、RHOA -Clausine E 和SPSB2 -CHEMBL175296)被提出。这些发现可能会促进我们对 ETV4 的新肿瘤基因关系的理解,并为其调节设计治疗策略。

"点击查看英文标题和摘要"
更新日期:2021-11-10

"点击查看英文标题和摘要"





















































 京公网安备 11010802027423号
京公网安备 11010802027423号