Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
DGL-LifeSci:生命科学图深度学习的开源工具包
ACS Omega ( IF 3.7 ) Pub Date : 2021-10-05 , DOI: 10.1021/acsomega.1c04017
Mufei Li 1 , Jinjing Zhou 1 , Jiajing Hu 2 , Wenxuan Fan 3 , Yangkang Zhang 4 , Yaxin Gu 3 , George Karypis 5, 6
ACS Omega ( IF 3.7 ) Pub Date : 2021-10-05 , DOI: 10.1021/acsomega.1c04017
Mufei Li 1 , Jinjing Zhou 1 , Jiajing Hu 2 , Wenxuan Fan 3 , Yangkang Zhang 4 , Yaxin Gu 3 , George Karypis 5, 6
Affiliation
|
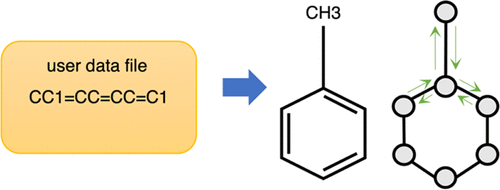
图神经网络(GNN)构成了一类图数据的深度学习方法。它们在化学和生物学中具有广泛的应用,例如分子性质预测、反应预测和药物-靶标相互作用预测。尽管有兴趣,基于 GNN 的建模仍然具有挑战性,因为除了编程和深度学习之外,它还需要图数据预处理和建模。在这里,我们展示了深度图库 (DGL)-LifeSci,这是一个用于生命科学图深度学习的开源包。Deep Graph Library (DGL)-LifeSci 是一个基于 RDKit、PyTorch 和 Deep Graph Library (DGL) 的 Python 工具包。DGL-LifeSci 允许在自定义数据集上进行基于 GNN 的建模,用于分子特性预测、反应预测和分子生成。凭借其命令行界面,用户无需任何编程和深度学习背景即可进行建模。我们使用标准基准测试 MoleculeNet、USPTO 和 ZINC 测试命令行界面。与之前的实现相比,DGL-LifeSci 实现了高达 6 倍的速度提升。为了建模灵活性,DGL-LifeSci 为建模管道的各个阶段提供了优化良好的模块。此外,DGL-LifeSci 提供了预训练模型,用于重现测试实验结果和应用无需训练的模型。该代码在 Apache-2.0 许可下分发,可在 https://github.com/awslabs/dgl-lifesci 上免费访问。DGL-LifeSci 为建模管道的各个阶段提供了优化良好的模块。此外,DGL-LifeSci 提供了预训练模型,用于重现测试实验结果和应用无需训练的模型。该代码在 Apache-2.0 许可下分发,可在 https://github.com/awslabs/dgl-lifesci 上免费访问。DGL-LifeSci 为建模管道的各个阶段提供了优化良好的模块。此外,DGL-LifeSci 提供了预训练模型,用于重现测试实验结果和应用无需训练的模型。该代码在 Apache-2.0 许可下分发,可在 https://github.com/awslabs/dgl-lifesci 上免费访问。

"点击查看英文标题和摘要"
更新日期:2021-10-19

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号