当前位置:
X-MOL 学术
›
Anal. Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
CRISPR Technique Incorporated with Single-Cell RNA Sequencing for Studying Hepatitis B Infection
Analytical Chemistry ( IF 6.7 ) Pub Date : 2021-07-30 , DOI: 10.1021/acs.analchem.1c02227
Connie Le 1 , Yanming Liu 2 , Joaquín López-Orozco 3 , Michael A Joyce 1 , X Chris Le 2 , D Lorne Tyrrell 1
Analytical Chemistry ( IF 6.7 ) Pub Date : 2021-07-30 , DOI: 10.1021/acs.analchem.1c02227
Connie Le 1 , Yanming Liu 2 , Joaquín López-Orozco 3 , Michael A Joyce 1 , X Chris Le 2 , D Lorne Tyrrell 1
Affiliation
|
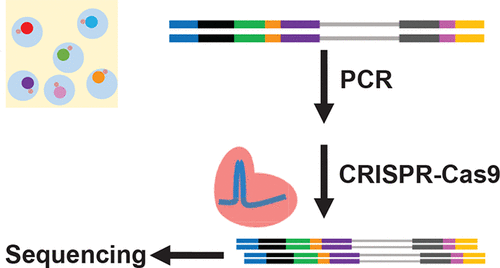
Single-cell RNA sequencing (scRNA-seq) provides rich transcriptomic information for studying molecular events and cell heterogeneity at the single-cell level. However, it is challenging to obtain sequence information from rare or low-abundance genes in the presence of other highly abundant genes. We report here a CRISPR-Cas9 technique for the depletion of high-abundance transcripts, resulting in preferential enrichment of rare transcripts. We demonstrate an application of this CRISPR-mediated enrichment technique to scRNA-seq of liver cells infected with hepatitis B virus (HBV). Direct sequencing without the CRISPR-mediated enrichment detected HBV RNA in only 0.6% of the cells. The CRISPR-mediated depletion of the three most abundant transcripts resulted in selective enrichment of the HBV transcript and successful sequencing of HBV RNA in more than 74% of the cells. The improvement enabled a study of HBV infection and interferon treatment of a liver cell model. Gene clusters between the control and HBV-infected Huh7.5-NTCP cells were similar, suggesting that HBV infection did not significantly alter gene expression of the host cells. The treatment with interferon alpha dramatically changed the gene expression of Huh7.5-NTCP cells. These results from the single cell RNA-seq analysis of 7370 cells are consistent with those of bulk experiments, suggesting that HBV is a “stealth virus”.
中文翻译:

结合单细胞 RNA 测序的 CRISPR 技术用于研究乙型肝炎感染
单细胞 RNA 测序 (scRNA-seq) 为研究单细胞水平的分子事件和细胞异质性提供了丰富的转录组信息。然而,在存在其他高丰度基因的情况下,从稀有或低丰度基因中获取序列信息具有挑战性。我们在此报告了一种用于去除高丰度转录本的 CRISPR-Cas9 技术,从而优先富集稀有转录本。我们展示了这种 CRISPR 介导的富集技术在感染乙型肝炎病毒 (HBV) 的肝细胞的 scRNA-seq 中的应用。没有 CRISPR 介导的富集的直接测序仅在 0.6% 的细胞中检测到 HBV RNA。CRISPR 介导的三种最丰富转录本的消耗导致 HBV 转录本的选择性富集和超过 74% 细胞中 HBV RNA 的成功测序。这一改进使得能够对肝细胞模型的 HBV 感染和干扰素治疗进行研究。对照和感染 HBV 的 Huh7.5-NTCP 细胞之间的基因簇相似,表明 HBV 感染没有显着改变宿主细胞的基因表达。用干扰素 α 处理显着改变了 Huh7.5-NTCP 细胞的基因表达。这些7370细胞单细胞RNA-seq分析的结果与批量实验的结果一致,表明HBV是一种“隐形病毒”。对照和感染 HBV 的 Huh7.5-NTCP 细胞之间的基因簇相似,表明 HBV 感染没有显着改变宿主细胞的基因表达。用干扰素 α 处理显着改变了 Huh7.5-NTCP 细胞的基因表达。这些7370细胞单细胞RNA-seq分析的结果与批量实验的结果一致,表明HBV是一种“隐形病毒”。对照和感染 HBV 的 Huh7.5-NTCP 细胞之间的基因簇相似,表明 HBV 感染没有显着改变宿主细胞的基因表达。用干扰素 α 处理显着改变了 Huh7.5-NTCP 细胞的基因表达。这些7370细胞单细胞RNA-seq分析的结果与批量实验的结果一致,表明HBV是一种“隐形病毒”。
更新日期:2021-08-10
中文翻译:

结合单细胞 RNA 测序的 CRISPR 技术用于研究乙型肝炎感染
单细胞 RNA 测序 (scRNA-seq) 为研究单细胞水平的分子事件和细胞异质性提供了丰富的转录组信息。然而,在存在其他高丰度基因的情况下,从稀有或低丰度基因中获取序列信息具有挑战性。我们在此报告了一种用于去除高丰度转录本的 CRISPR-Cas9 技术,从而优先富集稀有转录本。我们展示了这种 CRISPR 介导的富集技术在感染乙型肝炎病毒 (HBV) 的肝细胞的 scRNA-seq 中的应用。没有 CRISPR 介导的富集的直接测序仅在 0.6% 的细胞中检测到 HBV RNA。CRISPR 介导的三种最丰富转录本的消耗导致 HBV 转录本的选择性富集和超过 74% 细胞中 HBV RNA 的成功测序。这一改进使得能够对肝细胞模型的 HBV 感染和干扰素治疗进行研究。对照和感染 HBV 的 Huh7.5-NTCP 细胞之间的基因簇相似,表明 HBV 感染没有显着改变宿主细胞的基因表达。用干扰素 α 处理显着改变了 Huh7.5-NTCP 细胞的基因表达。这些7370细胞单细胞RNA-seq分析的结果与批量实验的结果一致,表明HBV是一种“隐形病毒”。对照和感染 HBV 的 Huh7.5-NTCP 细胞之间的基因簇相似,表明 HBV 感染没有显着改变宿主细胞的基因表达。用干扰素 α 处理显着改变了 Huh7.5-NTCP 细胞的基因表达。这些7370细胞单细胞RNA-seq分析的结果与批量实验的结果一致,表明HBV是一种“隐形病毒”。对照和感染 HBV 的 Huh7.5-NTCP 细胞之间的基因簇相似,表明 HBV 感染没有显着改变宿主细胞的基因表达。用干扰素 α 处理显着改变了 Huh7.5-NTCP 细胞的基因表达。这些7370细胞单细胞RNA-seq分析的结果与批量实验的结果一致,表明HBV是一种“隐形病毒”。







































 京公网安备 11010802027423号
京公网安备 11010802027423号