当前位置:
X-MOL 学术
›
J. Am. Chem. Soc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
用 DNA 重新定义蛋白质单晶内的蛋白质界面
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2021-06-05 , DOI: 10.1021/jacs.1c04191 Benjamin E Partridge 1 , Peter H Winegar 1 , Zhenyu Han 1 , Chad A Mirkin 1
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2021-06-05 , DOI: 10.1021/jacs.1c04191 Benjamin E Partridge 1 , Peter H Winegar 1 , Zhenyu Han 1 , Chad A Mirkin 1
Affiliation
|
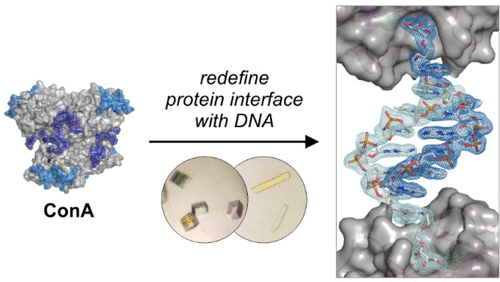
蛋白质是精致的纳米级构建模块:分子纯、化学可寻址,并且对其进化功能具有固有的选择性。将蛋白质组织成具有高位置、取向和平移顺序的单晶体会产生可以知道每个原子位置的材料。然而,由于定义天然单晶内蛋白质界面的无数相互作用,控制蛋白质的组织具有挑战性。最近,我们发现在蛋白质表面之间引入单一 DNA-DNA 相互作用会导致单晶体内蛋白质堆积的变化以及由此产生的蛋白质-蛋白质相互作用 (PPI)。然而,修改特定的 PPI 来对蛋白质包装进行有意的改变是一个尚未解决的挑战。在这项工作中,我们假设用 DNA-DNA 相互作用破坏和替换高度保守的 PPI 将使蛋白质包装能够通过利用引入的寡核苷酸的可编程性进行调节。使用刀豆球蛋白 A (ConA) 作为模型蛋白,我们规避了潜在有害的诱变,并利用 ConA 与甘露糖的选择性结合,将 DNA 非共价连接到蛋白质表面。我们证明 DNA 缔合消除了负责天然 ConA 结晶的主要 PPI,从而允许对 DNA 设计(长度、互补性和附着位置)进行微妙的改变,从而对 ConA 包装进行明显的改变,包括实现三种新颖的晶体结构和ConA 堆积沿单个晶轴的有意扩展。这些发现显着增强了我们对 DNA 如何取代天然 PPI 来编程有序材料中的蛋白质包装的理解。

"点击查看英文标题和摘要"
更新日期:2021-06-17

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号