当前位置:
X-MOL 学术
›
Microb. Biotechnol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
MiMiC:一种从宏基因组生成合成群落的生物信息学方法
Microbial Biotechnology ( IF 4.8 ) Pub Date : 2021-06-03 , DOI: 10.1111/1751-7915.13845
Neeraj Kumar 1, 2 , Thomas C A Hitch 1 , Dirk Haller 2, 3 , Ilias Lagkouvardos 2, 4 , Thomas Clavel 1
Affiliation
|
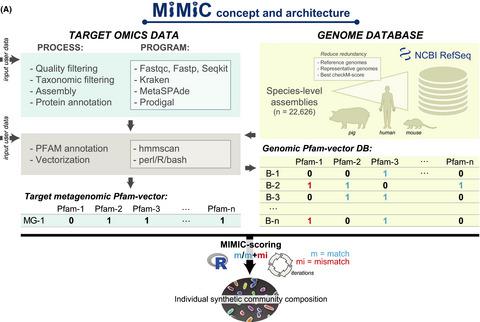
环境和宿主相关的微生物群落是复杂的生态系统,其中许多成员仍然未知。因此,研究群落动态具有挑战性,并且创建模仿主要群落功能的降低复杂性的模型系统很重要。因此,我们开发了 MiMiC,一种通过鸟枪法宏基因组进行数据驱动设计简化群落的计算方法。我们首先建立了物种级细菌和古菌基因组的综合数据库 ( n = 22 627),由蛋白质家族的二元(存在/不存在)载体 (Pfam = 17 929) 组成。 MiMiC 使用基于该数据库与任何输入宏基因组的 Pfam 二元向量之间的最大匹配与不匹配比的迭代评分系统来预测最小联合体的组成。 Pfam 矢量化保留了足够的分辨率来区分六个环境和宿主衍生的微生物群落之间的宏基因组图谱 ( n = 937)。计算得出的每个最小群落的物种数量在 5 到 11 之间,MiMiC 选择的群落比随机选择的物种更好地概括了原始样本的功能库。推断的最小群落保留了栖息地特定的特征,并且与由最丰富的成员组成的群落有很大不同。使用已知微生物的混合物揭示了从整个基因组数据库中选择 25 个目标物种中的 23 个的能力。 MiMiC 是开源的,可从 https://github.com/ClavelLab/MiMiC 获取。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号