当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Beyond Tripeptides Two-Step Active Machine Learning for Very Large Data sets
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2021-04-27 , DOI: 10.1021/acs.jctc.1c00159 Alexander van Teijlingen 1 , Tell Tuttle 1
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2021-04-27 , DOI: 10.1021/acs.jctc.1c00159 Alexander van Teijlingen 1 , Tell Tuttle 1
Affiliation
|
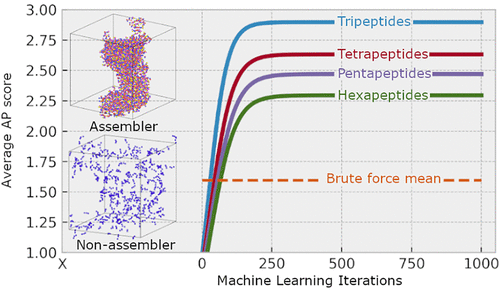
Self-assembling peptide nanostructures have been shown to be of great importance in nature and have presented many promising applications, for example, in medicine as drug-delivery vehicles, biosensors, and antivirals. Being very promising candidates for the growing field of bottom-up manufacture of functional nanomaterials, previous work (Frederix, et al. 2011 and 2015) has screened all possible amino acid combinations for di- and tripeptides in search of such materials. However, the enormous complexity and variety of linear combinations of the 20 amino acids make exhaustive simulation of all combinations of tetrapeptides and above infeasible. Therefore, we have developed an active machine-learning method (also known as “iterative learning” and “evolutionary search method”) which leverages a lower-resolution data set encompassing the whole search space and a just-in-time high-resolution data set which further analyzes those target peptides selected by the lower-resolution model. This model uses newly generated data upon each iteration to improve both lower- and higher-resolution models in the search for ideal candidates. Curation of the lower-resolution data set is explored as a method to control the selected candidates, based on criteria such as log P. A major aim of this method is to produce the best results in the least computationally demanding way. This model has been developed to be broadly applicable to other search spaces with minor changes to the algorithm, allowing its use in other areas of research.
中文翻译:

Beyond Tripeptides 针对超大数据集的两步主动机器学习
自组装肽纳米结构已被证明在自然界中具有重要意义,并已呈现出许多有前景的应用,例如,在医学中作为药物输送载体、生物传感器和抗病毒药物。作为功能性纳米材料自下而上制造领域非常有前途的候选者,之前的工作(Frederix 等人,2011 年和 2015 年)已经筛选了所有可能的二肽和三肽氨基酸组合,以寻找此类材料。然而,20 种氨基酸的线性组合的巨大复杂性和多样性使得对四肽及以上所有组合的详尽模拟是不可行的。所以,我们开发了一种主动机器学习方法(也称为“迭代学习”和“进化搜索方法”),它利用包含整个搜索空间的低分辨率数据集和实时高分辨率数据集进一步分析由较低分辨率模型选择的那些目标肽。该模型在每次迭代时使用新生成的数据来改进低分辨率和高分辨率模型,以寻找理想的候选者。较低分辨率数据集的管理被探索作为一种控制所选候选者的方法,基于诸如 log 之类的标准 该模型在每次迭代时使用新生成的数据来改进低分辨率和高分辨率模型,以寻找理想的候选者。较低分辨率数据集的管理被探索作为控制所选候选者的一种方法,基于诸如 log 之类的标准 该模型在每次迭代时使用新生成的数据来改进低分辨率和高分辨率模型,以寻找理想的候选者。较低分辨率数据集的管理被探索作为一种控制所选候选者的方法,基于诸如 log 之类的标准P。这种方法的一个主要目标是以最少的计算要求的方式产生最好的结果。该模型已被开发为广泛适用于其他搜索空间,只需对算法进行微小更改,即可将其用于其他研究领域。
更新日期:2021-05-11
中文翻译:

Beyond Tripeptides 针对超大数据集的两步主动机器学习
自组装肽纳米结构已被证明在自然界中具有重要意义,并已呈现出许多有前景的应用,例如,在医学中作为药物输送载体、生物传感器和抗病毒药物。作为功能性纳米材料自下而上制造领域非常有前途的候选者,之前的工作(Frederix 等人,2011 年和 2015 年)已经筛选了所有可能的二肽和三肽氨基酸组合,以寻找此类材料。然而,20 种氨基酸的线性组合的巨大复杂性和多样性使得对四肽及以上所有组合的详尽模拟是不可行的。所以,我们开发了一种主动机器学习方法(也称为“迭代学习”和“进化搜索方法”),它利用包含整个搜索空间的低分辨率数据集和实时高分辨率数据集进一步分析由较低分辨率模型选择的那些目标肽。该模型在每次迭代时使用新生成的数据来改进低分辨率和高分辨率模型,以寻找理想的候选者。较低分辨率数据集的管理被探索作为一种控制所选候选者的方法,基于诸如 log 之类的标准 该模型在每次迭代时使用新生成的数据来改进低分辨率和高分辨率模型,以寻找理想的候选者。较低分辨率数据集的管理被探索作为控制所选候选者的一种方法,基于诸如 log 之类的标准 该模型在每次迭代时使用新生成的数据来改进低分辨率和高分辨率模型,以寻找理想的候选者。较低分辨率数据集的管理被探索作为一种控制所选候选者的方法,基于诸如 log 之类的标准P。这种方法的一个主要目标是以最少的计算要求的方式产生最好的结果。该模型已被开发为广泛适用于其他搜索空间,只需对算法进行微小更改,即可将其用于其他研究领域。






























 京公网安备 11010802027423号
京公网安备 11010802027423号