当前位置:
X-MOL 学术
›
Anal. Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
An Ion Chromatography–Ultrahigh-Resolution-MS1/Data-Independent High-Resolution MS2 Method for Stable Isotope-Resolved Metabolomics Reconstruction of Central Metabolic Networks
Analytical Chemistry ( IF 6.7 ) Pub Date : 2021-01-22 , DOI: 10.1021/acs.analchem.0c03070
Qiushi Sun 1 , Teresa W-M Fan 1, 2, 3 , Andrew N Lane 1, 2, 3 , Richard M Higashi 1, 2, 3
Analytical Chemistry ( IF 6.7 ) Pub Date : 2021-01-22 , DOI: 10.1021/acs.analchem.0c03070
Qiushi Sun 1 , Teresa W-M Fan 1, 2, 3 , Andrew N Lane 1, 2, 3 , Richard M Higashi 1, 2, 3
Affiliation
|
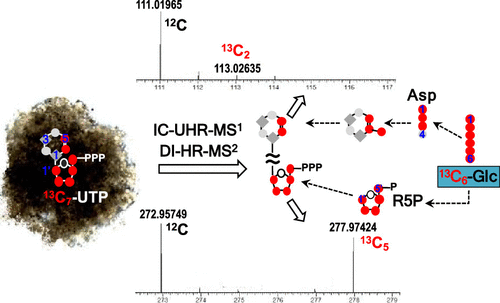
The metabolome comprises a complex network of interconnecting enzyme-catalyzed reactions that involve transfers of numerous molecular subunits. Thus, the reconstruction of metabolic networks requires metabolite substructures to be tracked. Subunit tracking can be achieved by tracing stable isotopes through metabolic transformations using NMR and ultrahigh -resolution (UHR)-mass spectrometry (MS). UHR-MS1 readily resolves and counts isotopic labels in metabolites but requires tandem MS to help identify isotopic enrichment in substructures. However, it is challenging to perform chromatography-based UHR-MS1 with its long acquisition time, while acquiring MS2 data on many coeluting labeled isotopologues for each metabolite. We have developed an ion chromatography (IC)-UHR-MS1/data-independent(DI)-HR-MS2 method to trace the fate of 13C atoms from [13C6]-glucose ([13C6]-Glc) in 3D A549 spheroids in response to anticancer selenite and simultaneously 13C/15N atoms from [13C5,15N2]-glutamine ([13C5,15N2]-Gln) in 2D BEAS-2B cells in response to arsenite transformation. This method retains the complete isotopologue distributions of metabolites via UHR-MS1 while simultaneously acquiring substructure label information via DI-MS2. These details in metabolite labeling patterns greatly facilitate rigorous reconstruction of multiple, intersecting metabolic pathways of central metabolism, which are illustrated here for the purine/pyrimidine nucleotide biosynthesis. The pathways reconstructed based on subunit-level isotopologue analysis further reveal specific enzyme-catalyzed reactions that are impacted by selenite or arsenite treatments.
中文翻译:

用于中心代谢网络的稳定同位素分辨代谢组学重建的离子色谱 - 超高分辨率 MS1/数据独立高分辨率 MS2 方法
代谢组包括相互连接的酶催化反应的复杂网络,其中涉及许多分子亚基的转移。因此,代谢网络的重建需要追踪代谢物亚结构。亚基追踪可以通过使用 NMR 和超高分辨率 (UHR)-质谱 (MS) 通过代谢转化追踪稳定同位素来实现。UHR-MS 1可以轻松解析和计算代谢物中的同位素标记,但需要串联 MS 来帮助识别子结构中的同位素富集。然而,在采集 MS 2的同时执行基于色谱的 UHR-MS 1具有挑战性,因为它的采集时间长每种代谢物的许多共流出标记同位素的数据。我们开发了一种离子色谱法 (IC)-UHR-MS 1 /数据独立 (DI)-HR-MS 2方法来追踪来自 [ 13 C 6 ]-葡萄糖 ([ 13 C 6 ]-的13 个C 原子的命运Glc) 在 3D A549 球体中响应抗癌亚硒酸盐和同时来自 [ 13 C 5 , 15 N 2 ]-谷氨酰胺 ([ 13 C 5 , 15 N 2 ) 的13 C/ 15 N 原子]-Gln) 在 2D BEAS-2B 细胞中响应亚砷酸盐转化。该方法通过 UHR-MS 1保留代谢物的完整同位素分布,同时通过 DI-MS 2获取子结构标签信息。代谢物标记模式中的这些细节极大地促进了对中枢代谢的多个交叉代谢途径的严格重建,此处对嘌呤/嘧啶核苷酸生物合成进行了说明。基于亚基水平同位素体分析重建的途径进一步揭示了受亚硒酸盐或亚砷酸盐处理影响的特定酶催化反应。
更新日期:2021-02-09
中文翻译:

用于中心代谢网络的稳定同位素分辨代谢组学重建的离子色谱 - 超高分辨率 MS1/数据独立高分辨率 MS2 方法
代谢组包括相互连接的酶催化反应的复杂网络,其中涉及许多分子亚基的转移。因此,代谢网络的重建需要追踪代谢物亚结构。亚基追踪可以通过使用 NMR 和超高分辨率 (UHR)-质谱 (MS) 通过代谢转化追踪稳定同位素来实现。UHR-MS 1可以轻松解析和计算代谢物中的同位素标记,但需要串联 MS 来帮助识别子结构中的同位素富集。然而,在采集 MS 2的同时执行基于色谱的 UHR-MS 1具有挑战性,因为它的采集时间长每种代谢物的许多共流出标记同位素的数据。我们开发了一种离子色谱法 (IC)-UHR-MS 1 /数据独立 (DI)-HR-MS 2方法来追踪来自 [ 13 C 6 ]-葡萄糖 ([ 13 C 6 ]-的13 个C 原子的命运Glc) 在 3D A549 球体中响应抗癌亚硒酸盐和同时来自 [ 13 C 5 , 15 N 2 ]-谷氨酰胺 ([ 13 C 5 , 15 N 2 ) 的13 C/ 15 N 原子]-Gln) 在 2D BEAS-2B 细胞中响应亚砷酸盐转化。该方法通过 UHR-MS 1保留代谢物的完整同位素分布,同时通过 DI-MS 2获取子结构标签信息。代谢物标记模式中的这些细节极大地促进了对中枢代谢的多个交叉代谢途径的严格重建,此处对嘌呤/嘧啶核苷酸生物合成进行了说明。基于亚基水平同位素体分析重建的途径进一步揭示了受亚硒酸盐或亚砷酸盐处理影响的特定酶催化反应。

































 京公网安备 11010802027423号
京公网安备 11010802027423号