当前位置:
X-MOL 学术
›
Biochemistry
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Mechanism of Deoxyguanosine Diphosphate Insertion by Human DNA Polymerase β
Biochemistry ( IF 2.9 ) Pub Date : 2021-01-21 , DOI: 10.1021/acs.biochem.0c00847 Fausto A Varela 1 , Bret D Freudenthal 1
Biochemistry ( IF 2.9 ) Pub Date : 2021-01-21 , DOI: 10.1021/acs.biochem.0c00847 Fausto A Varela 1 , Bret D Freudenthal 1
Affiliation
|
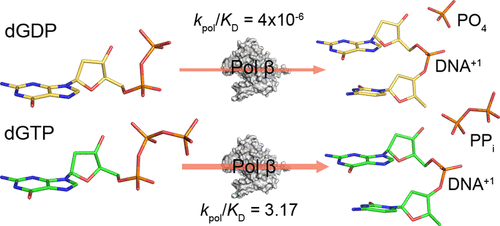
DNA polymerases play vital roles in the maintenance and replication of genomic DNA by synthesizing new nucleotide polymers using nucleoside triphosphates as substrates. Deoxynucleoside triphosphates (dNTPs) are the canonical substrates for DNA polymerases; however, some bacterial polymerases have been demonstrated to insert deoxynucleoside diphosphates (dNDPs), which lack a third phosphate group, the γ-phosphate. Whether eukaryotic polymerases can efficiently incorporate dNDPs has not been investigated, and much about the chemical or structural role played by the γ-phosphate of dNTPs remains unknown. Using the model mammalian polymerase (Pol) β, we examine how Pol β incorporates a substrate lacking a γ-phosphate [deoxyguanosine diphosphate (dGDP)] utilizing kinetic and crystallographic approaches. Using single-turnover kinetics, we determined dGDP insertion across a templating dC by Pol β to be drastically impaired when compared to dGTP insertion. We found the most significant impairment in the apparent insertion rate (kpol), which was reduced 32000-fold compared to that of dGTP insertion. X-ray crystal structures revealed similar enzyme–substrate contacts for both dGDP and dGTP. These findings suggest the insertion efficiency of dGDP is greatly decreased due to impairments in polymerase chemistry. This work is the first instance of a mammalian polymerase inserting a diphosphate nucleotide and provides insight into the nature of polymerase mechanisms by highlighting how these enzymes have evolved to use triphosphate nucleotide substrates.
中文翻译:

人 DNA 聚合酶 β 插入脱氧鸟苷二磷酸的机制
DNA 聚合酶通过使用核苷三磷酸作为底物合成新的核苷酸聚合物,在基因组 DNA 的维持和复制中发挥重要作用。脱氧核苷三磷酸 (dNTP) 是 DNA 聚合酶的典型底物;然而,一些细菌聚合酶已被证明可以插入脱氧核苷二磷酸 (dNDP),它缺少第三个磷酸基,即 γ-磷酸。真核聚合酶是否可以有效地掺入 dNDPs 尚未得到研究,而且关于 dNTPs 的 γ-磷酸所起的化学或结构作用的许多信息仍然未知。使用模型哺乳动物聚合酶 (Pol) β,我们研究了 Pol β 如何利用动力学和晶体学方法结合缺乏 γ-磷酸 [脱氧鸟苷二磷酸 (dGDP)] 的底物。使用单周转动力学,与 dGTP 插入相比,我们确定通过 Pol β 在模板 dC 中插入 dGDP 会严重受损。我们发现表观插入率最显着的损伤(k pol ), 与 dGTP 插入相比减少了 32000 倍。X 射线晶体结构显示 dGDP 和 dGTP 具有相似的酶-底物接触。这些发现表明,由于聚合酶化学受损,dGDP 的插入效率大大降低。这项工作是哺乳动物聚合酶插入二磷酸核苷酸的第一个实例,并通过强调这些酶如何进化为使用三磷酸核苷酸底物来深入了解聚合酶机制的性质。
更新日期:2021-02-09
中文翻译:

人 DNA 聚合酶 β 插入脱氧鸟苷二磷酸的机制
DNA 聚合酶通过使用核苷三磷酸作为底物合成新的核苷酸聚合物,在基因组 DNA 的维持和复制中发挥重要作用。脱氧核苷三磷酸 (dNTP) 是 DNA 聚合酶的典型底物;然而,一些细菌聚合酶已被证明可以插入脱氧核苷二磷酸 (dNDP),它缺少第三个磷酸基,即 γ-磷酸。真核聚合酶是否可以有效地掺入 dNDPs 尚未得到研究,而且关于 dNTPs 的 γ-磷酸所起的化学或结构作用的许多信息仍然未知。使用模型哺乳动物聚合酶 (Pol) β,我们研究了 Pol β 如何利用动力学和晶体学方法结合缺乏 γ-磷酸 [脱氧鸟苷二磷酸 (dGDP)] 的底物。使用单周转动力学,与 dGTP 插入相比,我们确定通过 Pol β 在模板 dC 中插入 dGDP 会严重受损。我们发现表观插入率最显着的损伤(k pol ), 与 dGTP 插入相比减少了 32000 倍。X 射线晶体结构显示 dGDP 和 dGTP 具有相似的酶-底物接触。这些发现表明,由于聚合酶化学受损,dGDP 的插入效率大大降低。这项工作是哺乳动物聚合酶插入二磷酸核苷酸的第一个实例,并通过强调这些酶如何进化为使用三磷酸核苷酸底物来深入了解聚合酶机制的性质。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号