当前位置:
X-MOL 学术
›
Mol. Ther. Nucl. Acids
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
设计合成 circRNA 的高效反向剪接和翻译
Molecular Therapy - Nucleic Acids ( IF 6.5 ) Pub Date : 2021-01-16 , DOI: 10.1016/j.omtn.2021.01.003 Rita M. Meganck , Jiacheng Liu , Andrew E. Hale , Katherine E. Simon , Marco M. Fanous , Heather A. Vincent , Jeremy E. Wilusz , Nathaniel J. Moorman , William F. Marzluff , Aravind Asokan
Molecular Therapy - Nucleic Acids ( IF 6.5 ) Pub Date : 2021-01-16 , DOI: 10.1016/j.omtn.2021.01.003 Rita M. Meganck , Jiacheng Liu , Andrew E. Hale , Katherine E. Simon , Marco M. Fanous , Heather A. Vincent , Jeremy E. Wilusz , Nathaniel J. Moorman , William F. Marzluff , Aravind Asokan
|
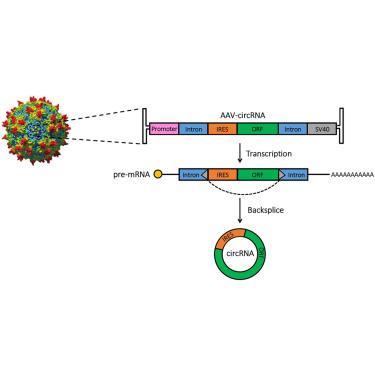
环状 RNA (circRNA) 是高度稳定的 RNA 分子,是治疗性蛋白质和非编码 RNA 表达的有吸引力的模板。在真核生物中,circRNA 主要由剪接体通过反向剪接产生。在这里,我们询问不同的分子元素,包括内含子类型和长度、重复、内部核糖体进入位点 (IRES) 和 circRNA 形成所必需的外显子长度,并利用这些信息来设计强大的反向剪接和 circRNA 表达。具体来说,我们利用下游内含子可以耐受大插入而不影响剪接的发现来实现来自同一模板的反向剪接的 circRNA 和 tRNA 内含子 circRNA 的串联表达。此外,截断选定的内含子区域显着增加了不同细胞类型中的 circRNA 形成以及心脏和骨骼肌组织中 AAV 介导的 circRNA 表达。我们还观察到不同的 IRES 元件和外显子长度影响 circRNA 的表达和翻译,揭示了外显子对剪接的贡献,正如产生的不同 RNA 种类所证明的那样。总而言之,这些数据为改进合成 circRNA 的设计和表达提供了新的见解。当与 AAV 衣壳和启动子技术相结合时,构成该模块化平台的反向剪接内含子和 IRES 元件显着扩展了基因表达工具包。

"点击查看英文标题和摘要"
更新日期:2021-01-16

"点击查看英文标题和摘要"































 京公网安备 11010802027423号
京公网安备 11010802027423号