当前位置:
X-MOL 学术
›
Biochemistry
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
人类DNA失配修复蛋白沿DNA的预识别扩散机制:Msh2-Msh3与Msh2-Msh6
Biochemistry ( IF 2.9 ) Pub Date : 2020-12-15 , DOI: 10.1021/acs.biochem.0c00669 Arumay Pal 1 , Harry M. Greenblatt 1 , Yaakov Levy 1
Biochemistry ( IF 2.9 ) Pub Date : 2020-12-15 , DOI: 10.1021/acs.biochem.0c00669 Arumay Pal 1 , Harry M. Greenblatt 1 , Yaakov Levy 1
Affiliation
|
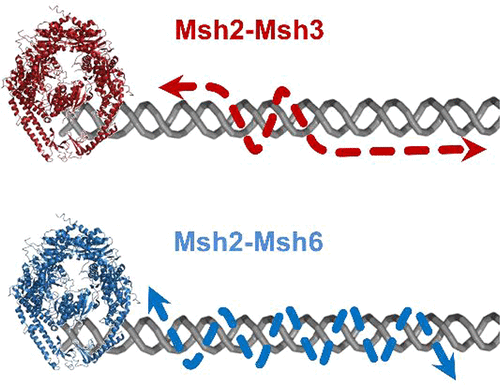
DNA错配修复(MMR)是重要的复制后过程,可消除错配或未配对的核苷酸,以确保基因组复制保真度。在人类中,Msh2-Msh6和Msh2-Msh3是识别DNA损伤的两个错配修复起始因子。尽管这些蛋白质的X射线晶体结构与DNA损伤复合存在,但在沿非特异性双链DNA的初始搜索过程中对其结构了解甚少,因为它们寿命短且难以通过实验确定。在这项研究中,各种计算方法被用来规避这些困难。基于Msh2-Msh3和Msh2-Msh6晶体结构的全原子和粗粒度模拟显示沿DNA没有翻译,这表明初始搜索构象与病变结合的晶体结构不同。我们对MSH蛋白质的可能搜索模式结构进行了建模,并使用粗粒度分子动力学模拟显示了它们可以在DNA上进行旋转耦合扩散,这是一种对其功能适当且有效的搜索机制,并且通过荧光共振可以较早地预测出一种能量转移和荧光显微镜研究。这种搜索机制是通过错配结合结构域(MBD),钳位结构域和DNA主链之间的静电相互作用实现的。在模拟过程中,它们的扩散速率不会随盐浓度的增加而显着变化,这与来自实验研究的观察结果一致。当它们的DNA结合钳之间的间隙增加时,Msh2-Msh3主要通过钳域扩散,而Msh2-Msh6仍使用MBD扩散,再现实验测量的Msh2-Msh6较低的扩散系数。有趣的是,Msh2-Msh3能够从DNA解离,而Msh2-Msh6总是在DNA双链体上扩散。这与Msh2-Msh3与Msh2-Msh6不同,可以克服诸如核小体等障碍的实验观察结果一致。我们的模型提供了由Msh2-Msh6和Msh2-Msh3进行的不同错配搜索机制的分子图,尽管它们的结构相似。

"点击查看英文标题和摘要"
更新日期:2020-12-29

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号