当前位置:
X-MOL 学术
›
Anal. Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Enhanced Collision Induced Unfolding and Electron Capture Dissociation of Native-like Protein Ions
Analytical Chemistry ( IF 6.7 ) Pub Date : 2020-11-09 , DOI: 10.1021/acs.analchem.0c03372
Varun V Gadkari 1 , Carolina Rojas Ramírez 1 , Daniel D Vallejo 1 , Ruwan T Kurulugama 2 , John C Fjeldsted 2 , Brandon T Ruotolo 1
Analytical Chemistry ( IF 6.7 ) Pub Date : 2020-11-09 , DOI: 10.1021/acs.analchem.0c03372
Varun V Gadkari 1 , Carolina Rojas Ramírez 1 , Daniel D Vallejo 1 , Ruwan T Kurulugama 2 , John C Fjeldsted 2 , Brandon T Ruotolo 1
Affiliation
|
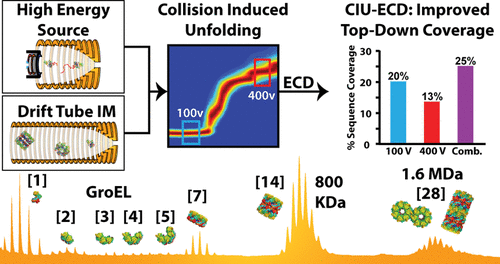
Native ion mobility-mass spectrometry (IM-MS) is capable of revealing much that remains unknown within the structural proteome, promising such information on refractory protein targets. Here, we report the development of a unique drift tube IM-MS (DTIM-MS) platform, which combines high-energy source optics for improved collision induced unfolding (CIU) experiments and an electromagnetostatic cell for electron capture dissociation (ECD). We measured a series of high precision collision cross section (CCS) values for protein and protein complex ions ranging from 6–1600 kDa, exhibiting an average relative standard deviation (RSD) of 0.43 ± 0.20%. Furthermore, we compare our CCS results to previously reported DTIM values, finding strong agreement across similarly configured instrumentation (average RSD of 0.82 ± 0.73%), and systematic differences for DTIM CCS values commonly used to calibrate traveling-wave IM separators (−3% average RSD). Our CIU experiments reveal that the modified DTIM-MS instrument described here achieves enhanced levels of ion activation when compared with any previously reported IM-MS platforms, allowing for comprehensive unfolding of large multiprotein complex ions as well as interplatform CIU comparisons. Using our modified DTIM instrument, we studied two protein complexes. The enhanced CIU capabilities enable us to study the gas phase stability of the GroEL 7-mer and 14-mer complexes. Finally, we report CIU-ECD experiments for the alcohol dehydrogenase tetramer, demonstrating improved sequence coverage by combining ECD fragmentation integrated over multiple CIU intermediates. Further improvements for such native top-down sequencing experiments were possible by leveraging IM separation, which enabled us to separate and analyze CID and ECD fragmentation simultaneously.
中文翻译:

增强类天然蛋白离子的碰撞诱导展开和电子捕获解离
天然离子淌度质谱 (IM-MS) 能够揭示结构蛋白质组中许多未知的信息,有望提供有关难降解蛋白质靶点的此类信息。在这里,我们报告了独特的漂移管 IM-MS (DTIM-MS) 平台的开发,该平台结合了用于改进碰撞诱导展开 (CIU) 实验的高能源光学器件和用于电子捕获解离 (ECD) 的静电磁池。我们测量了一系列 6-1600 kDa 范围内的蛋白质和蛋白质复合离子的高精度碰撞截面 (CCS) 值,显示平均相对标准偏差 (RSD) 为 0.43 ± 0.20%。此外,我们将我们的 CCS 结果与之前报告的 DTIM 值进行比较,发现类似配置的仪器之间存在很强的一致性(平均 RSD 为 0.82 ± 0.73%),并且通常用于校准行波 IM 分离器的 DTIM CCS 值存在系统差异(−3%平均 RSD)。我们的 CIU 实验表明,与之前报道的任何 IM-MS 平台相比,此处描述的改进的 DTIM-MS 仪器实现了更高水平的离子激活,从而可以全面展开大型多蛋白复合离子以及平台间 CIU 比较。使用我们改进的 DTIM 仪器,我们研究了两种蛋白质复合物。增强的 CIU 功能使我们能够研究 GroEL 7 聚体和 14 聚体复合物的气相稳定性。最后,我们报告了醇脱氢酶四聚体的 CIU-ECD 实验,证明通过将 ECD 片段整合到多个 CIU 中间体上,可以提高序列覆盖率。通过利用 IM 分离,可以进一步改进此类天然自上而下测序实验,这使我们能够同时分离和分析 CID 和 ECD 碎片。
更新日期:2020-12-01
中文翻译:

增强类天然蛋白离子的碰撞诱导展开和电子捕获解离
天然离子淌度质谱 (IM-MS) 能够揭示结构蛋白质组中许多未知的信息,有望提供有关难降解蛋白质靶点的此类信息。在这里,我们报告了独特的漂移管 IM-MS (DTIM-MS) 平台的开发,该平台结合了用于改进碰撞诱导展开 (CIU) 实验的高能源光学器件和用于电子捕获解离 (ECD) 的静电磁池。我们测量了一系列 6-1600 kDa 范围内的蛋白质和蛋白质复合离子的高精度碰撞截面 (CCS) 值,显示平均相对标准偏差 (RSD) 为 0.43 ± 0.20%。此外,我们将我们的 CCS 结果与之前报告的 DTIM 值进行比较,发现类似配置的仪器之间存在很强的一致性(平均 RSD 为 0.82 ± 0.73%),并且通常用于校准行波 IM 分离器的 DTIM CCS 值存在系统差异(−3%平均 RSD)。我们的 CIU 实验表明,与之前报道的任何 IM-MS 平台相比,此处描述的改进的 DTIM-MS 仪器实现了更高水平的离子激活,从而可以全面展开大型多蛋白复合离子以及平台间 CIU 比较。使用我们改进的 DTIM 仪器,我们研究了两种蛋白质复合物。增强的 CIU 功能使我们能够研究 GroEL 7 聚体和 14 聚体复合物的气相稳定性。最后,我们报告了醇脱氢酶四聚体的 CIU-ECD 实验,证明通过将 ECD 片段整合到多个 CIU 中间体上,可以提高序列覆盖率。通过利用 IM 分离,可以进一步改进此类天然自上而下测序实验,这使我们能够同时分离和分析 CID 和 ECD 碎片。

































 京公网安备 11010802027423号
京公网安备 11010802027423号