当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
PRosettaC: Rosetta Based Modeling of PROTAC Mediated Ternary Complexes
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2020-09-25 , DOI: 10.1021/acs.jcim.0c00589 Daniel Zaidman 1 , Jaime Prilusky 2 , Nir London 1
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2020-09-25 , DOI: 10.1021/acs.jcim.0c00589 Daniel Zaidman 1 , Jaime Prilusky 2 , Nir London 1
Affiliation
|
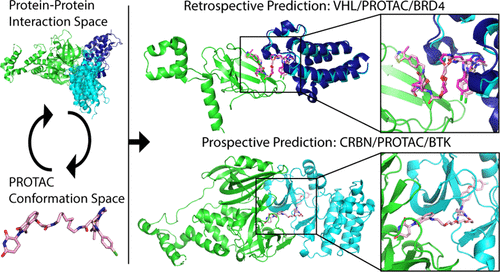
Proteolysis-targeting chimeras (PROTACs), which induce degradation by recruitment of an E3 ligase to a target protein, are gaining much interest as a new pharmacological modality. However, designing PROTACs is challenging. Formation of a ternary complex between the protein target, the PROTAC, and the recruited E3 ligase is considered paramount for successful degradation. A structural model of this ternary complex could in principle inform rational PROTAC design. Unfortunately, only a handful of structures are available for such complexes, necessitating tools for their modeling. We developed a combined protocol for the modeling of a ternary complex induced by a given PROTAC. Our protocol alternates between sampling of the protein–protein interaction space and the PROTAC molecule conformational space. Application of this protocol—PRosettaC—to a benchmark of known PROTAC ternary complexes results in near-native predictions, with often atomic accuracy prediction of the protein chains, as well as the PROTAC binding moieties. It allowed the modeling of a CRBN/BTK complex that recapitulated experimental results for a series of PROTACs. PRosettaC generated models may be used to design PROTACs for new targets, as well as improve PROTACs for existing targets, potentially cutting down time and synthesis efforts. To enable wide access to this protocol, we have made it available through a web server (https://prosettac.weizmann.ac.il/).
中文翻译:

PROsettaC:基于 Rosetta 的 PROTAC 介导的三元复合物建模
蛋白水解靶向嵌合体 (PROTAC) 通过将 E3 连接酶募集到靶蛋白上来诱导降解,作为一种新的药理学方式引起了人们的广泛关注。然而,设计 PROTAC 具有挑战性。蛋白质靶标、PROTAC 和招募的 E3 连接酶之间形成三元复合物被认为对于成功降解至关重要。这种三元复合物的结构模型原则上可以为合理的 PROTAC 设计提供信息。不幸的是,只有少数结构可用于此类复合体,因此需要使用工具进行建模。我们开发了一个组合协议,用于对给定 PROTAC 诱导的三元复合物进行建模。我们的协议在蛋白质-蛋白质相互作用空间和 PROTAC 分子构象空间的采样之间交替。将此协议(PROsettaC)应用于已知 PROTAC 三元复合物的基准会产生接近天然的预测,通常对蛋白质链以及 PROTAC 结合部分进行原子精度预测。它允许对 CRBN/BTK 复合物进行建模,概括一系列 PROTAC 的实验结果。 PROsettaC 生成的模型可用于设计新目标的 PROTAC,以及改进现有目标的 PROTAC,从而可能减少时间和合成工作。为了实现对该协议的广泛访问,我们通过网络服务器提供了它(https://prosettac.weizmann.ac.il/)。
更新日期:2020-10-26
中文翻译:

PROsettaC:基于 Rosetta 的 PROTAC 介导的三元复合物建模
蛋白水解靶向嵌合体 (PROTAC) 通过将 E3 连接酶募集到靶蛋白上来诱导降解,作为一种新的药理学方式引起了人们的广泛关注。然而,设计 PROTAC 具有挑战性。蛋白质靶标、PROTAC 和招募的 E3 连接酶之间形成三元复合物被认为对于成功降解至关重要。这种三元复合物的结构模型原则上可以为合理的 PROTAC 设计提供信息。不幸的是,只有少数结构可用于此类复合体,因此需要使用工具进行建模。我们开发了一个组合协议,用于对给定 PROTAC 诱导的三元复合物进行建模。我们的协议在蛋白质-蛋白质相互作用空间和 PROTAC 分子构象空间的采样之间交替。将此协议(PROsettaC)应用于已知 PROTAC 三元复合物的基准会产生接近天然的预测,通常对蛋白质链以及 PROTAC 结合部分进行原子精度预测。它允许对 CRBN/BTK 复合物进行建模,概括一系列 PROTAC 的实验结果。 PROsettaC 生成的模型可用于设计新目标的 PROTAC,以及改进现有目标的 PROTAC,从而可能减少时间和合成工作。为了实现对该协议的广泛访问,我们通过网络服务器提供了它(https://prosettac.weizmann.ac.il/)。

































 京公网安备 11010802027423号
京公网安备 11010802027423号