当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
使用分子动力学模拟对固有紊乱的蛋白质进行小分子调节。
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2020-08-03 , DOI: 10.1021/acs.jcim.0c00381
Pablo Herrera-Nieto 1 , Adrià Pérez 1 , Gianni De Fabritiis 1, 2, 3
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2020-08-03 , DOI: 10.1021/acs.jcim.0c00381
Pablo Herrera-Nieto 1 , Adrià Pérez 1 , Gianni De Fabritiis 1, 2, 3
Affiliation
|
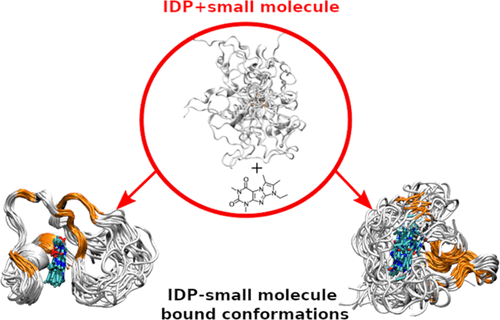
本质上无序的蛋白质的极端动态行为阻碍了能够调节它们的类药物化合物的发展。有几个与无序肽特异性相互作用的小分子的例子。但是,它们的作用机理仍未得到很好的理解。在这里,我们将广泛的分子动力学模拟与自适应采样算法结合使用,以在固有无序蛋白的情况下进行自由配体结合研究。我们在由无序蛋白p27的D2子域和小分子SJ403组成的系统中测试了该方法。结果显示了几种蛋白-配体结合状态,其特征是通过p27的配体与关键残基之间有限数量的接触介导了松散定向的相互作用。最后,分离的蛋白质也可能探索处于结合状态的蛋白质构象,因此支持了其中添加小分子限制了可用构象空间的模型。

"点击查看英文标题和摘要"
更新日期:2020-08-03

"点击查看英文标题和摘要"







































 京公网安备 11010802027423号
京公网安备 11010802027423号