当前位置:
X-MOL 学术
›
J. Am. Chem. Soc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Modelling DNA-strand displacement reactions in the presence of base-pair mismatches
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2020-06-04 , DOI: 10.1021/jacs.0c03105 Patrick Irmisch 1 , Thomas E Ouldridge 2 , Ralf Seidel 1
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2020-06-04 , DOI: 10.1021/jacs.0c03105 Patrick Irmisch 1 , Thomas E Ouldridge 2 , Ralf Seidel 1
Affiliation
|
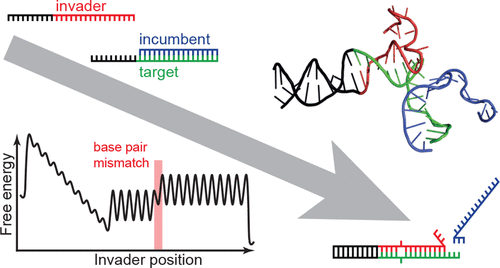
Toehold-mediated strand displacement is the most abundantly used method to achieve dynamic switching in DNA-based nanotechnology. An 'invader' strand binds to the 'toehold' overhang of a target strand and replaces a target-bound 'incumbent' strand. Hereby, complementarity of the invader to the single-stranded toehold provides the energetic bias of the reaction. Despite the widespread use of strand displacement reactions for realizing dynamic DNA nanostructures, variants on the basic motif have not been completely characterized. Here we introduce a simple thermodynamic model, which is capable of quantitatively describing the kinetics of strand displacement reactions in the presence of mismatches, using a minimal set of parameters. Furthermore, our model highlights that base pair fraying and internal loop formation are important mechanisms when involving mismatches in the displacement process. Our model should provide a helpful tool for the rational design of strand-displacement reaction networks.
中文翻译:

在存在碱基对错配的情况下模拟 DNA 链置换反应
Toehold 介导的链置换是在基于 DNA 的纳米技术中实现动态转换的最常用的方法。“入侵者”链与目标链的“立足点”突出端结合,并取代与目标结合的“现有”链。因此,入侵者与单链立足点的互补性提供了反应的能量偏差。尽管广泛使用链置换反应来实现动态 DNA 纳米结构,但尚未完全表征基本基序上的变体。在这里,我们引入了一个简单的热力学模型,该模型能够使用最少的参数集定量描述存在错配时链置换反应的动力学。此外,我们的模型强调,在置换过程中涉及错配时,碱基对磨损和内部环形成是重要的机制。我们的模型应该为链置换反应网络的合理设计提供有用的工具。
更新日期:2020-06-04
中文翻译:

在存在碱基对错配的情况下模拟 DNA 链置换反应
Toehold 介导的链置换是在基于 DNA 的纳米技术中实现动态转换的最常用的方法。“入侵者”链与目标链的“立足点”突出端结合,并取代与目标结合的“现有”链。因此,入侵者与单链立足点的互补性提供了反应的能量偏差。尽管广泛使用链置换反应来实现动态 DNA 纳米结构,但尚未完全表征基本基序上的变体。在这里,我们引入了一个简单的热力学模型,该模型能够使用最少的参数集定量描述存在错配时链置换反应的动力学。此外,我们的模型强调,在置换过程中涉及错配时,碱基对磨损和内部环形成是重要的机制。我们的模型应该为链置换反应网络的合理设计提供有用的工具。































 京公网安备 11010802027423号
京公网安备 11010802027423号