当前位置:
X-MOL 学术
›
J. Am. Chem. Soc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Ultrafast DNA sensors with DNA framework-bridged hybridization reactions
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2020-05-05 , DOI: 10.1021/jacs.9b13737 Fengqin Li 1, 2 , Xiuhai Mao 1 , Fan Li 1 , Min Li 1 , Jianlei Shen 1 , Zhilei Ge 1 , Chunhai Fan 1 , Xiaolei Zuo 1
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2020-05-05 , DOI: 10.1021/jacs.9b13737 Fengqin Li 1, 2 , Xiuhai Mao 1 , Fan Li 1 , Min Li 1 , Jianlei Shen 1 , Zhilei Ge 1 , Chunhai Fan 1 , Xiaolei Zuo 1
Affiliation
|
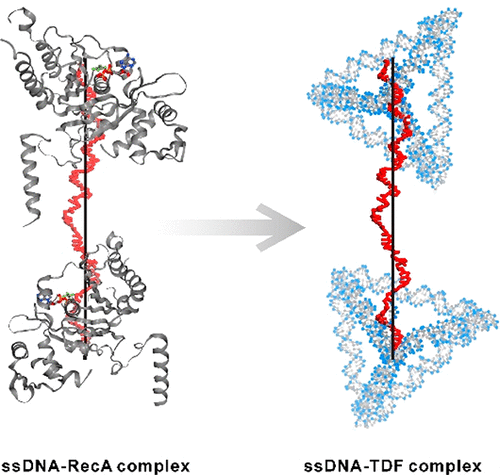
Intracellular DNA-based hybridization reactions generally undergo under tension rather than in free states, which are spa-tiotemporally controlled in physiological conditions. However, how nanomechanical forces affect DNA hybridization effi-ciencies in in-vitro DNA assays, e.g. biosensors or biochips, remains largely elusive. Here we design DNA framework-based nanomechanical handles that can control the stretching states of DNA molecules. Using a pair of tetrahedral DNA frame-work (TDF) nanostructured handles, we develop bridge DNA sensors that can capture target DNA with ultrafast speed and high efficiency. We find that the rigid TDF handles binds two ends of a single-stranded DNA (ssDNA) and holds it at a stretched state, with apparent stretching length comparing to its counterpart of double-stranded DNA (dsDNA) via atom-ic force microscopy (AFM) measurement. The DNA stretching effect of ssDNA is then monitored using single molecular fluorescence energy transfer (FRET), resulting in decreased FRET efficiency in the stretched ssDNA. By controlling the stretching state of ssDNA, we obtained significantly improved hybridization kinetics (within 1 min) and hybridization effi-ciency (~ 98%) under the target concentration of 500 nM. The bridge DNA sensors demonstrated high sensitivity (1 fM), high specificity (single mismatch mutation discrimination) and high selectivity (suitable for the detection in serum and blood) under the target concentration of 10 nM. The controlling of stretching state of ssDNA show great potential in the applications of biosensors, bioimaging and biochips.
中文翻译:

具有 DNA 框架桥接杂交反应的超快 DNA 传感器
基于细胞内 DNA 的杂交反应通常在张力下而不是在自由状态下进行,自由状态是在生理条件下时空控制的。然而,纳米力学力如何影响体外 DNA 分析(例如生物传感器或生物芯片)中的 DNA 杂交效率,在很大程度上仍然难以捉摸。在这里,我们设计了基于 DNA 框架的纳米机械手柄,可以控制 DNA 分子的拉伸状态。使用一对四面体 DNA 框架 (TDF) 纳米结构手柄,我们开发了能够以超快速度和高效率捕获目标 DNA 的桥式 DNA 传感器。我们发现刚性 TDF 手柄结合单链 DNA (ssDNA) 的两端并将其保持在拉伸状态,通过原子力显微镜 (AFM) 测量与双链 DNA (dsDNA) 的对应物相比,具有表观拉伸长度。然后使用单分子荧光能量转移 (FRET) 监测 ssDNA 的 DNA 拉伸效应, 导致拉伸 ssDNA 中的 FRET 效率降低。通过控制 ssDNA 的拉伸状态,我们在 500 nM 的目标浓度下获得了显着改善的杂交动力学(1 分钟内)和杂交效率(~ 98%)。桥式 DNA 传感器在 10 nM 的目标浓度下表现出高灵敏度 (1 fM)、高特异性(单一错配突变识别)和高选择性(适用于血清和血液中的检测)。ssDNA拉伸状态的控制在生物传感器的应用中显示出巨大的潜力,
更新日期:2020-05-05
中文翻译:

具有 DNA 框架桥接杂交反应的超快 DNA 传感器
基于细胞内 DNA 的杂交反应通常在张力下而不是在自由状态下进行,自由状态是在生理条件下时空控制的。然而,纳米力学力如何影响体外 DNA 分析(例如生物传感器或生物芯片)中的 DNA 杂交效率,在很大程度上仍然难以捉摸。在这里,我们设计了基于 DNA 框架的纳米机械手柄,可以控制 DNA 分子的拉伸状态。使用一对四面体 DNA 框架 (TDF) 纳米结构手柄,我们开发了能够以超快速度和高效率捕获目标 DNA 的桥式 DNA 传感器。我们发现刚性 TDF 手柄结合单链 DNA (ssDNA) 的两端并将其保持在拉伸状态,通过原子力显微镜 (AFM) 测量与双链 DNA (dsDNA) 的对应物相比,具有表观拉伸长度。然后使用单分子荧光能量转移 (FRET) 监测 ssDNA 的 DNA 拉伸效应, 导致拉伸 ssDNA 中的 FRET 效率降低。通过控制 ssDNA 的拉伸状态,我们在 500 nM 的目标浓度下获得了显着改善的杂交动力学(1 分钟内)和杂交效率(~ 98%)。桥式 DNA 传感器在 10 nM 的目标浓度下表现出高灵敏度 (1 fM)、高特异性(单一错配突变识别)和高选择性(适用于血清和血液中的检测)。ssDNA拉伸状态的控制在生物传感器的应用中显示出巨大的潜力,


















































 京公网安备 11010802027423号
京公网安备 11010802027423号