Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
PreDBA:一种用于预测蛋白质-DNA结合亲和力的异质集成方法。
Scientific Reports ( IF 3.8 ) Pub Date : 2020-01-28 , DOI: 10.1038/s41598-020-57778-1 Wenyi Yang 1 , Lei Deng 1, 2
Scientific Reports ( IF 3.8 ) Pub Date : 2020-01-28 , DOI: 10.1038/s41598-020-57778-1 Wenyi Yang 1 , Lei Deng 1, 2
Affiliation
|
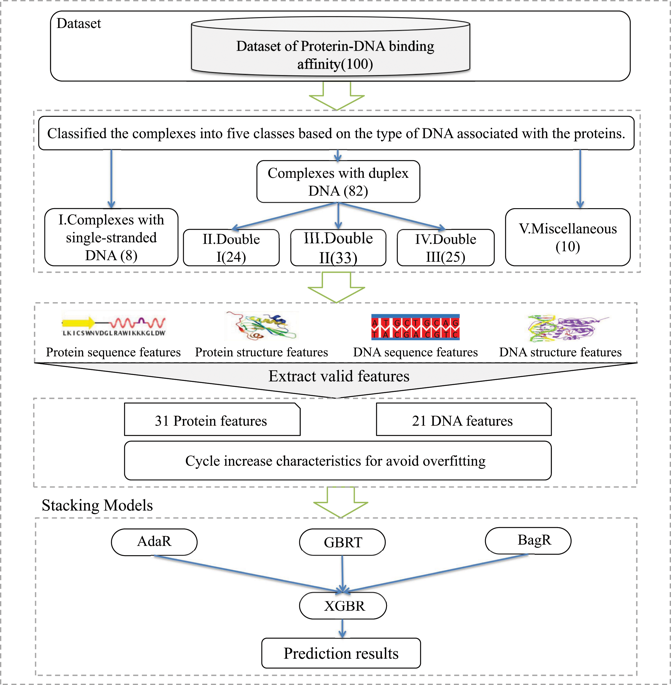
蛋白质与DNA之间的相互作用在各种关键的自然过程中起着至关重要的作用,例如DNA复制,转录,剪接和修复。研究蛋白质与DNA的结合亲和力有助于了解蛋白质-DNA复合物的识别机制。由于通过实验测量的蛋白质-DNA结合亲和力数据仍然存在许多局限性,因此需要准确而可靠的计算方法。因此,我们在本文中提出了一种称为PreDBA的计算方法,该方法可以通过使用异类集成模型有效地预测蛋白质与DNA的结合亲和力。从相关文献中手动收集了一百种蛋白质-DNA复合物,作为蛋白质-DNA结合亲和力的数据集。然后,获得52个序列和结构特征。基于此,计算出这52个特征与蛋白质-DNA结合亲和力之间的相关性。此外,我们发现蛋白质与DNA的结合亲和力受化合物DNA分子结构的影响。根据与构成蛋白质-DNA复合物的蛋白质相关的DNA结构,我们将所有蛋白质-DNA化合物分为五类。在每个组中,基于获得的特征构建堆叠的异类集成模型。最后,基于绑定亲和力数据集,我们使用了留一法交叉验证对所提出的方法进行了全面评估。在这五个类别中,我们推荐的方法的Pearson相关系数值在0.735至0.926之间。

"点击查看英文标题和摘要"
更新日期:2020-01-29

"点击查看英文标题和摘要"















































 京公网安备 11010802027423号
京公网安备 11010802027423号