当前位置:
X-MOL 学术
›
ACS Chem. Biol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
An Evolved RNA Recognition Motif That Suppresses HIV-1 Tat/TAR-Dependent Transcription
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2016-06-13 00:00:00 , DOI: 10.1021/acschembio.6b00145
David W. Crawford , Brett D. Blakeley , Po-Han Chen 1 , Chringma Sherpa 2 , Stuart F.J. Le Grice 2 , Ite A. Laird-Offringa 1 , Brian R. McNaughton
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2016-06-13 00:00:00 , DOI: 10.1021/acschembio.6b00145
David W. Crawford , Brett D. Blakeley , Po-Han Chen 1 , Chringma Sherpa 2 , Stuart F.J. Le Grice 2 , Ite A. Laird-Offringa 1 , Brian R. McNaughton
Affiliation
|
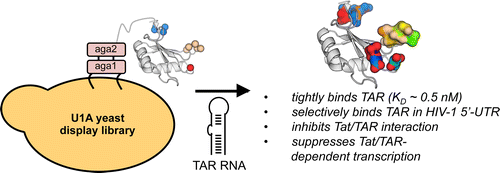
Potent and selective recognition and modulation of disease-relevant RNAs remain a daunting challenge. We previously examined the utility of the U1A N-terminal RNA recognition motif as a scaffold for tailoring new RNA hairpin recognition and showed that as few as one or two mutations can result in moderate affinity (low μM dissociation constant) for the human immunodeficiency virus (HIV) trans-activation response element (TAR) RNA, an RNA hairpin controlling transcription of the human immunodeficiency virus (HIV) genome. Here, we use yeast display and saturation mutagenesis of established RNA-binding regions in U1A to identify new synthetic proteins that potently and selectively bind TAR RNA. Our best candidate has truly altered, not simply broadened, RNA-binding selectivity; it binds TAR with subnanomolar affinity (apparent dissociation constant of ∼0.5 nM) but does not appreciably bind the original U1A RNA target (U1hpII). It specifically recognizes the TAR RNA hairpin in the context of the HIV-1 5′-untranslated region, inhibits the interaction between TAR RNA and an HIV trans-activator of transcription (Tat)-derived peptide, and suppresses Tat/TAR-dependent transcription. Proteins described in this work are among the tightest TAR RNA-binding reagents–small molecule, nucleic acid, or protein–reported to date and thus have potential utility as therapeutics and basic research tools. Moreover, our findings demonstrate how a naturally occurring RNA recognition motif can be dramatically resurfaced through mutation, leading to potent and selective recognition—and modulation—of disease-relevant RNA.
中文翻译:

抑制HIV-1 Tat / TAR依赖转录的进化的RNA识别母题。
与疾病相关的RNA的有效,选择性识别和调节仍然是一项艰巨的挑战。我们先前检查了U1A N端RNA识别基序作为用于定制新RNA发夹识别的支架的效用,并显示少至一个或两个突变可导致对人免疫缺陷病毒的中等亲和力(低μM解离常数)( HIV)反式激活应答元件(TAR)RNA,一种控制人类免疫缺陷病毒(HIV)基因组转录的RNA发夹。在这里,我们使用酵母展示和U1A中已建立的RNA结合区的饱和诱变来鉴定有效并选择性结合TAR RNA的新合成蛋白。我们的最佳人选确实改变了而不是简单地扩大了RNA结合的选择性。它以亚纳摩尔亲和力(表观解离常数约0.5 nM)与TAR结合,但与原始U1A RNA靶(U1hpII)没有明显结合。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。5 nM),但未明显结合原始U1A RNA靶标(U1hpII)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。5 nM),但未明显结合原始U1A RNA靶标(U1hpII)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道的最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有作为治疗剂和基础研究工具的潜在用途。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。并抑制Tat / TAR依赖的转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。并抑制Tat / TAR依赖的转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。
更新日期:2016-06-13
中文翻译:

抑制HIV-1 Tat / TAR依赖转录的进化的RNA识别母题。
与疾病相关的RNA的有效,选择性识别和调节仍然是一项艰巨的挑战。我们先前检查了U1A N端RNA识别基序作为用于定制新RNA发夹识别的支架的效用,并显示少至一个或两个突变可导致对人免疫缺陷病毒的中等亲和力(低μM解离常数)( HIV)反式激活应答元件(TAR)RNA,一种控制人类免疫缺陷病毒(HIV)基因组转录的RNA发夹。在这里,我们使用酵母展示和U1A中已建立的RNA结合区的饱和诱变来鉴定有效并选择性结合TAR RNA的新合成蛋白。我们的最佳人选确实改变了而不是简单地扩大了RNA结合的选择性。它以亚纳摩尔亲和力(表观解离常数约0.5 nM)与TAR结合,但与原始U1A RNA靶(U1hpII)没有明显结合。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。5 nM),但未明显结合原始U1A RNA靶标(U1hpII)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。5 nM),但未明显结合原始U1A RNA靶标(U1hpII)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。它在HIV-1 5'非翻译区的背景下特异性识别TAR RNA发夹,抑制TAR RNA与HIV反转录激活因子(Tat)衍生的肽之间的相互作用,并抑制Tat / TAR依赖性转录。这项工作中描述的蛋白质是迄今为止报道的最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有作为治疗剂和基础研究工具的潜在用途。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。并抑制Tat / TAR依赖的转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。并抑制Tat / TAR依赖的转录。这项工作中描述的蛋白质是迄今为止报道最紧密的TAR RNA结合试剂(小分子,核酸或蛋白质)之一,因此具有潜在的用途,可作为治疗剂和基础研究工具。此外,我们的发现证明了天然存在的RNA识别基序如何能够通过突变显着地重新出现,从而导致与疾病相关的RNA的有效和选择性识别(以及调节)。

































 京公网安备 11010802027423号
京公网安备 11010802027423号