当前位置:
X-MOL 学术
›
J. Am. Chem. Soc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
通过 DNA 杂交探针识别单个核苷酸变体的工程化动力学扩增机制
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2016-04-11 , DOI: 10.1021/jacs.6b00277 Sherry Xi Chen 1 , Georg Seelig 1, 2
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2016-04-11 , DOI: 10.1021/jacs.6b00277 Sherry Xi Chen 1 , Georg Seelig 1, 2
Affiliation
|
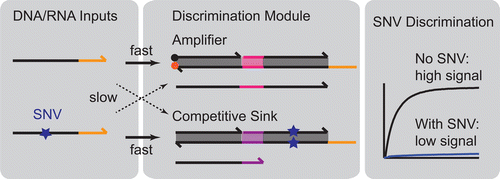
即使是两个完全相同的生物核酸序列之间的单核苷酸差异也会产生显着的功能后果。在这里,我们使用模型引导的反应途径工程来定量提高选择性杂交探针在识别单核苷酸变体 (SNV) 方面的性能。具体来说,我们构建了一个检测系统,该系统将竞争歧视与基于 DNA 链置换的催化扩增相结合。我们在数学上和实验上都表明,这种系统在与单链 DNA 和 RNA 结合方面的单核苷酸选择性比单独由于竞争性杂交引起的歧视好二次方。作为一个额外的好处,集成电路继承了放大的特性,并提供比标准杂交探针至少高 10 倍的灵敏度。此外,我们展示了如何调整检测机制,使得检测反应与目标序列中 SNV 的位置无关。相比之下,先前设计用于动力学辨别的基于链置换的探针对位置效应高度敏感。我们应用我们的系统来可靠地区分 let-7 microRNA 家族的不同成员,这些成员仅在单个碱基位置上有所不同。我们的结果证明了系统反应网络设计在定量改进生物技术方面的力量。我们展示了如何调整检测机制,使得检测反应与目标序列中 SNV 的位置无关。相比之下,先前设计用于动力学辨别的基于链置换的探针对位置效应高度敏感。我们应用我们的系统来可靠地区分 let-7 microRNA 家族的不同成员,这些成员仅在单个碱基位置上有所不同。我们的结果证明了系统反应网络设计在定量改进生物技术方面的力量。我们展示了如何调整检测机制,使得检测反应与目标序列中 SNV 的位置无关。相比之下,先前设计用于动力学辨别的基于链置换的探针对位置效应高度敏感。我们应用我们的系统来可靠地区分 let-7 microRNA 家族的不同成员,这些成员仅在单个碱基位置上有所不同。我们的结果证明了系统反应网络设计在定量改进生物技术方面的力量。我们应用我们的系统来可靠地区分 let-7 microRNA 家族的不同成员,这些成员仅在单个碱基位置上有所不同。我们的结果证明了系统反应网络设计在定量改进生物技术方面的力量。我们应用我们的系统来可靠地区分 let-7 microRNA 家族的不同成员,这些成员仅在单个碱基位置上有所不同。我们的结果证明了系统反应网络设计在定量改进生物技术方面的力量。

"点击查看英文标题和摘要"
更新日期:2016-04-11

"点击查看英文标题和摘要"

















































 京公网安备 11010802027423号
京公网安备 11010802027423号