浙江大学王勇课题组JCTC | 开发新型机器学习驱动的自适应采样算法,提升生物分子模拟效率
英文原题:Enhanced Sampling of Biomolecular Slow Conformational Transitions Using Adaptive Sampling and Machine Learning 通讯作者:王勇(浙江大学);吴昊(上海交通大学) 作者:张鸣远, 吴昊*, 王勇*
近日,浙江大学生命科学学院王勇研究员团队联合上海交通大学吴昊教授团队,成功开发了一种创新的算法,结合自适应采样与机器学习技术,显著提升了生物分子动力学模拟中捕捉慢构象转变过程的能力。这一研究成果表明,在计算资源有限的情况下,机器学习算法在增强生物分子复杂运动过程模拟中的潜力。
分子动力学模拟作为结构生物学和药物化学的重要工具,能提供分子层面的独特洞察。然而,由于现有计算硬件和算法的限制,传统分子动力学模拟在时间尺度和构象空间采样方面存在瓶颈。增强采样算法通过添加偏置势加速采样过程,但依赖于能否正确选择集合变量(Collective Variables),而如何在复杂生物系统中确定最优集合变量始终是一个难题。
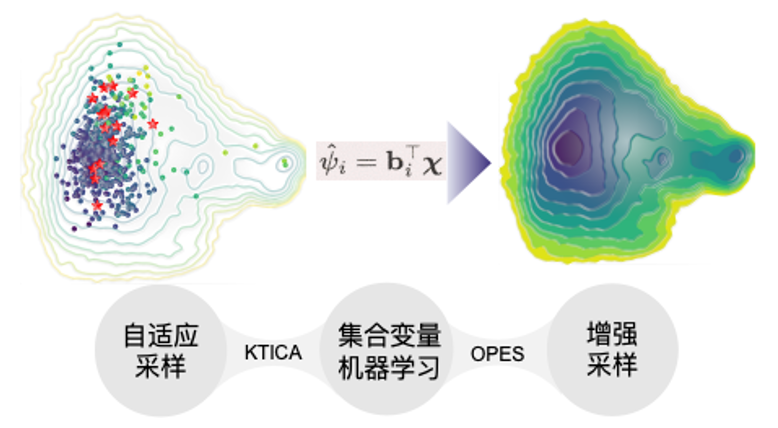
图1. 算法思路示意图
该研究提出了一种将自适应采样与库普曼重加权滞后时间独立分量分析(KTICA)和即时概率增强采样(OPES)相结合的新方法(图1)。这一算法无需预先了解体系的详细信息,能够自动探索体系的势能面、学习集合变量并进行高效的增强采样。在对两个多肽模型的测试中,该方法不仅准确捕捉了具有物理意义的集合变量,还能自动检测自适应采样的收敛情况,并将收敛的集合变量传递给OPES算法,最终快速获得收敛的自由能地貌。
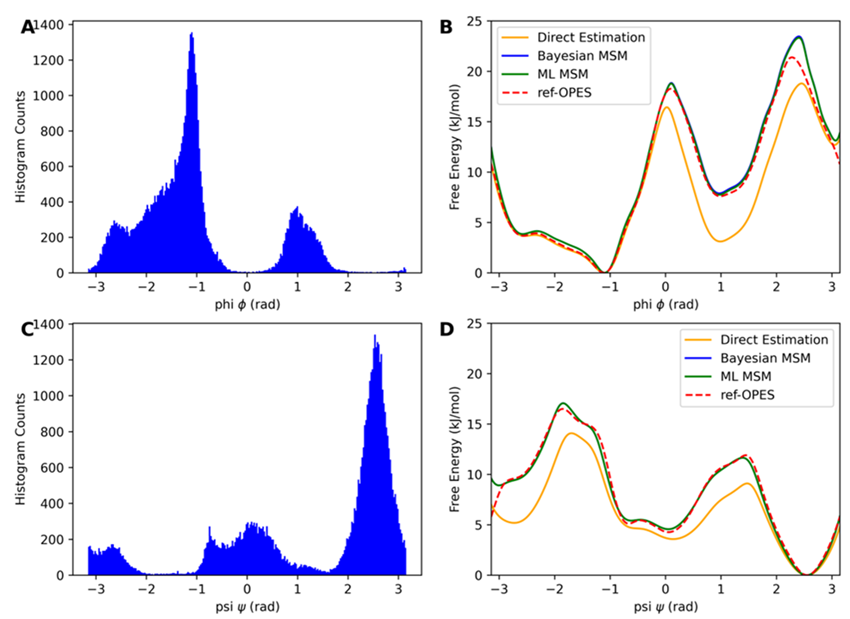
图2. 相比传统的马尔可夫模型的自由能计算方法,新算法在相同的模拟时长下表现出更高的效率与精度
相比传统的马尔可夫模型的自由能计算方法,新算法在相同的模拟时长下表现出更高的效率与精度,尤其在估算高能区能量壁垒时优势明显(图2)。研究表明,结合自适应采样与机器学习算法,有望突破增强采样中集合变量选择的瓶颈,为模拟更复杂的生物系统提供新的思路。
本研究由浙江大学生命科学学院王勇研究员和上海交通大学吴昊教授共同指导,张鸣远为第一作者。该工作得到了国家自然科学基金和国家“十四五”重点研发计划“生物与信息融合”专项的支持。
相关算法已开源,代码可访问:
https://github.com/Mingyuan00/Adaptive_Sampling_for_OPES
https://www.plumed-nest.org/eggs/24/019/
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Enhanced Sampling of Biomolecular Slow Conformational Transitions Using Adaptive Sampling and Machine Learning
Mingyuan Zhang, Hao Wu*, Yong Wang*
J. Chem. Theory Comput. 2024, 20, 19, 8569–8582
https://doi.org/10.1021/acs.jctc.4c00764
Published September 20, 2024
© 2024 American Chemical Society
(本稿件来自ACS Publications)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!
































 京公网安备 11010802027423号
京公网安备 11010802027423号