山东大学合作Angew:自抗性基因导向的脂肪酸合成酶抑制剂cerulenin的生物合成
Cerulenin(1)是首个天然来源的脂肪酸合成酶(FAS)抑制剂,它由2015年诺贝尔生理或医学奖得主大村智(Ōmura Satoshi)从真菌Cephalosporium caerulens(后被修正为Sarocladium oryzae)的发酵液中提取分离得到,具有显著的抗细菌、抗真菌和抗肿瘤活性。Cerulenin结构中含有一个亲水的顺式环氧基团和一个疏水的7E,10E-壬二烯基尾部;在质子溶液中,可以发生分子内5-exo-trig环化反应(图1)。2008年,Grininger课题组解析了cerulenin和酿酒酵母FAS α亚基的共晶结构,发现该分子的C-2/C-3环氧基团开环后与FAS α亚基酮脂酰合成酶结构域(KS domain)中第1305位的半胱氨酸巯基共价结合,从而抑制FAS活性(图1)。2024年,Burkart课题组通过同位素标记结合质谱和核磁实验,揭示了cerulenin与FAS半胱氨酸结合时发生了C-2/C-3逆醛醇键断裂(retro-aldol bond cleavage)和分子重排反应。此外,研究表明cerulenin可以减少小鼠进食量,增加其能量消耗,具有与瘦素leptin相似的减肥效果,因此被作为减肥药开发;但因其存在给药后体重急剧下降和厌食症等全身性不良反应,其临床应用受到限制。Cerulenin也是拓扑异构酶I抑制剂,促进肿瘤细胞凋亡,同时它作为探针分子被广泛应用于细胞生物学研究。
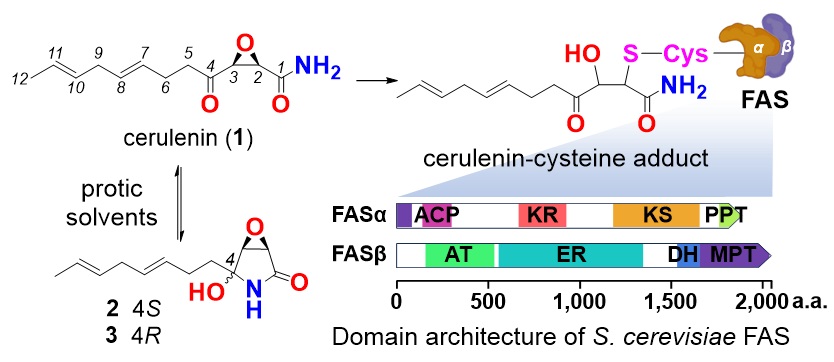
图1. Cerulenin的化学结构及其通过与酿酒酵母FASα亚基结合发挥抗真菌活性示意图。图片来源:Angew. Chem. Int. Ed.
尽管cerulenin的化学结构和生物活性被广泛研究,但cerulenin的生物合成从未被报道,仅有个别文献推测cerulenin源于脂肪酸合成途径。有意思的是,自cerulenin于上世纪60年代被发现以来,从未有结构类似物被报道,属于“孤儿分子”。近日,山东大学药学院尚卓课题组、西澳大学Yit Heng Chooi课题组和麦考瑞大学Andrew Piggott课题组合作在Angew. Chem. Int. Ed.杂志上首次报道了明星分子cerulenin的生物合成途径。
作者首先采用LCMS分析了S. attenuatum MST-FP2084和S. oryzae MST-FP2085的发酵提取物,确定这两株Sarocladium属真菌可以生产cerulenin (1)。从含有1% (w/v) XAD-16树脂的酪蛋白甘油培养基发酵液中分离得到了化合物1,并通过NMR鉴定了化合物1的化学结构。为了研究1的生物合成,作者对这两株真菌进行了全基因组测序、组装和生物合成基因簇预测。分析化合物1的化学结构,作者推测其生物合成基因簇可能编码包含高度还原的I型聚酮合酶(HR-PKS)、酰胺转移酶、细胞色素P450氧化酶等基因。考虑到1可以与真菌FAS共价结合产生抗真菌活性,作者推测cerulenin的生产菌株可能进化出“自抗性基因”以抵消cerulenin对自身的毒性,“自抗性基因”为真菌FAS α亚基的同源基因。为了从16个预测的PKS基因簇中快速定位cerulenin基因簇,作者通过生物信息学分析从两株真菌中寻找到了编码FAS的管家基因(包含FAS α亚基和β亚基),然后根据FAS α亚基的序列,从MST-FP2084的scaffold 176和MST-FP2085的scaffold 144上找到了编码FAS α亚基自抗性蛋白的“自抗性基因”,与管家基因的相似度为65%。更重要的是,两株菌的“FASα自抗性基因”上游都有一个包含11个基因的HR-PKS生物合成基因簇(命名为cer),推测其最有可能负责cerulenin的生物合成。接下来,作者对cer基因簇中的各个基因进行了功能注释,其中HR-PKS基因(cerA)编码多结构域的I型PKS,包括聚酮合酶(KS)、酰基转移酶(AT)、脱水酶(DH)、烯酰还原酶(ER)、酮还原酶(KR)和酰基载体蛋白(ACP)结构域。MST-FP2084和MST-FP2085菌株中cer基因簇具有99%的同源性(图2A)。随后,作者预测了位于cerA和cerI之间的基因所编码的蛋白质功能,包括谷氨酰胺转移酶(CerD)、硫酯酶(CerB)、2个氧化还原酶(CerC和CerE)、FAD依赖的氧化酶(CerF)、2个细胞色素P450单加氧酶(CerP1和CerP2)、细胞色素b5(CerJ)、转运蛋白(CerG)以及一个功能未知蛋白(CerH)(图2A)。另外,HR-PKS基因下游还有一个基因orf13编码一个单加氧酶。
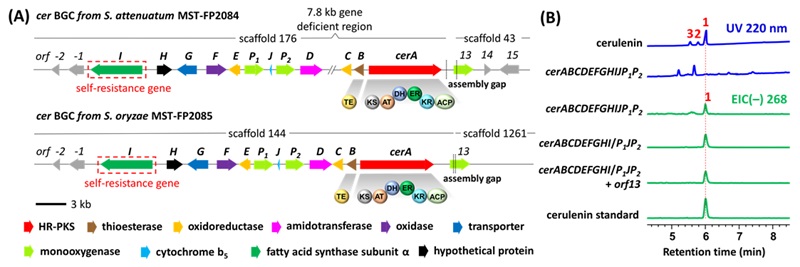
图2. cer基因簇的鉴定和验证(A:cer基因簇的基因注释;B:表达cer基因簇的Aspergillus nidulans LO8030发酵物的LC-MS分析)。图片来源:Angew. Chem. Int. Ed.
为了验证cer基因簇是否负责1的生物合成,作者在构巢曲霉Aspergillus nidulans LO8030中重构了cer的生物合成基因簇,将自抗性基因cerI与簇内其他基因共表达,在培养时添加XAD-16树脂以吸附并稳定化合物1。LCMS分析结果显示,组合cerABCDEFGHIJP1P2可生产化合物1,而加入基因orf13后并未检测到除1以外的新产物,表明cerABCDEFGHIJP1P2负责1的生物合成,且基因orf13不参与1的生物合成(图2B)。为了进一步阐明1的生物合成途径,作者构建了包含不同cer基因组合的过表达质粒,转入A. nidulans LO8030中进行异源表达。首先,从cerAB共表达菌株的乙酸乙酯提取物中鉴定了C8:3不饱和脂肪酸6、八元环聚酮7和具有6/4环结构的化合物 8-9,推测7-9是由C12:5(4)和C10:4(5)多不饱和脂肪酸通过自发电环化反应形成(图3B)。为了寻找cerAB共表达的真正产物,作者将XAD-16树脂加入到表达cerAB的构巢曲霉培养基中。作者从树脂的丙酮洗脱液中分离得到了主产物4,它和1都具有12个碳原子,同时也检测到了5和6(图3A),这表明电环化产物是自发形成的分流产物。尽管不稳定,作者仍然通过HRMS和NMR阐明了4的结构,结构中含有4个顺式双键和1个反式双键。
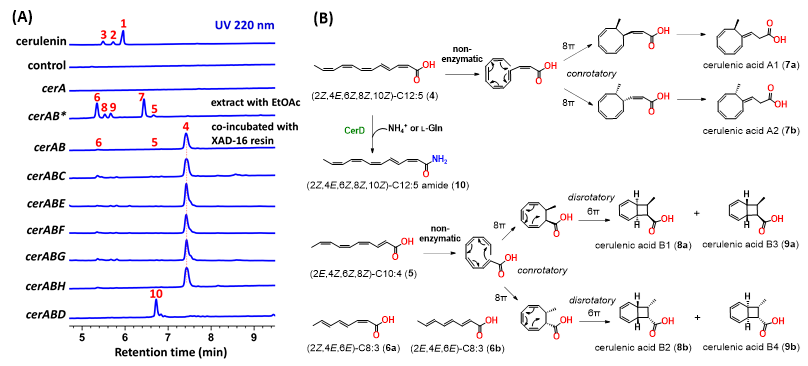
图3. 聚酮中间体4和5的异源表达和非酶转化。图片来源:Angew. Chem. Int. Ed.
为了阐明化合物4下游的生物合成途径,作者将cerAB和其他基因逐一共表达,发现只有共表达cerABD组合时可产生新的中间体多烯酰胺(10)(图3A),其绝对构型和4保持一致,这表明了基因cerD催化脂肪酸酰胺化。作者通过体外酶反应,进一步证明了无机铵离子和L-谷氨酰胺可作为CerD催化的酰胺化反应的氮供体(图5A)。接下来,cerABD与其他基因逐一共表达时没有检测到任何新的中间体生成。但有意思的是,共表达cerABDJP2或cerABDJP1P2导致前体10消失,作者推测CerP2和CerJ可能催化下一步反应,但由于产物的不稳定性导致LCMS未能检测到任何产物。因此,作者改变了研究策略,通过从完整的cer基因簇中逐个删除基因来研究每个基因的功能(图4A)。对含有cerABCDEFJP1P2(∆cerGHI)和cerABCDEFGIJP1P2(∆cerH)基因组合的A. nidulans LO8030树脂提取物进行LC-MS分析,均检测到1的存在,表明转运蛋白CerG、自抗性蛋白CerI和假设蛋白CerH不直接参与1的生物合成。另外,作者在∆cerGHI发酵提取物中还分离得4个cerulenin类似物(11−14),通过HRMS和NMR鉴定了结构,推测是构巢曲霉宿主还原∆2双键后形成的分流产物(图4B)。∆cerD菌株累积了含有五元内酯结构的中间体19和20,逆生物合成分析推测19和20是化合物4经过CerP2J催化的Δ4环氧化和自发开环酯化产生(图4B)。为了确定CerP1或CerP2是否参与Δ4环氧化,作者对表达完整cer基因簇但缺少cerP1、cerP2、cerJ或其组合的构巢曲霉发酵提取物进行了LC-MS分析。结果表明,在ΔcerP2、ΔcerJ、ΔcerJP1、ΔcerJP2和ΔcerJP1P2菌株中积累了多烯酰胺10,而ΔcerP1菌株产生12,推测是由C-4/C-5环氧化物中间体产生的分流产物。∆cerEF菌株的主产物为中间体15,次要产物包括化合物16−18。作者推断17和18是由C-4/C-5环氧酰胺(Ⅰ)中间体经过CerC催化的双键迁移和CerP1J催化的∆2,3环氧化反应得到的(图4B)。
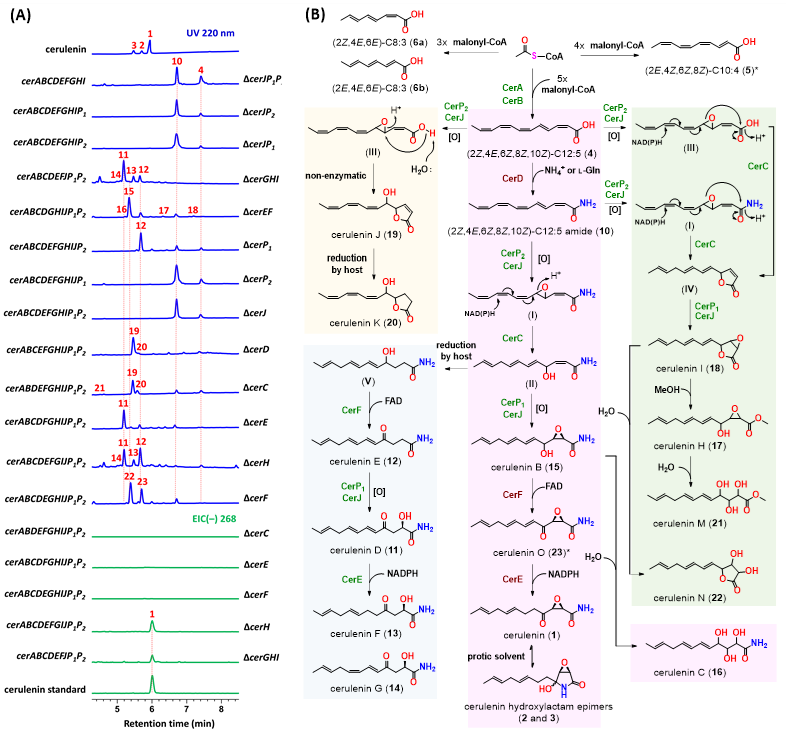
图4. 不同cer基因组合在构巢曲霉中的异源表达,以及基于体内体外实验构建的cerulenin(1)生物合成途径。图片来源:Angew. Chem. Int. Ed.
接下来,作者推测CerF可能催化关键生物合成中间体15的4位羟基氧化为酮,CerE还原∆5双键,生成终产物1。由于FAD依赖的氧化酶CerF去除信号肽后失去催化活性,氧化还原酶CerE可能为膜蛋白,于是作者将15与表达cerF的构巢曲霉细胞裂解液共孵育,通过LC-MS分析检测到化合物23的生成,证实了CerF具有氧化4位羟基为酮基的功能(图5)。然后,将15与共表达cerEF的构巢曲霉细胞裂解液反应,检测到终产物1的生成,证实了CerE作为一种烯酰还原酶,具有还原∆5双键的功能(图5)。通过构建多基因缺失组合,作者共分离鉴定了cerulenin生物合成中间体或者副产物18个,均为新化合物。
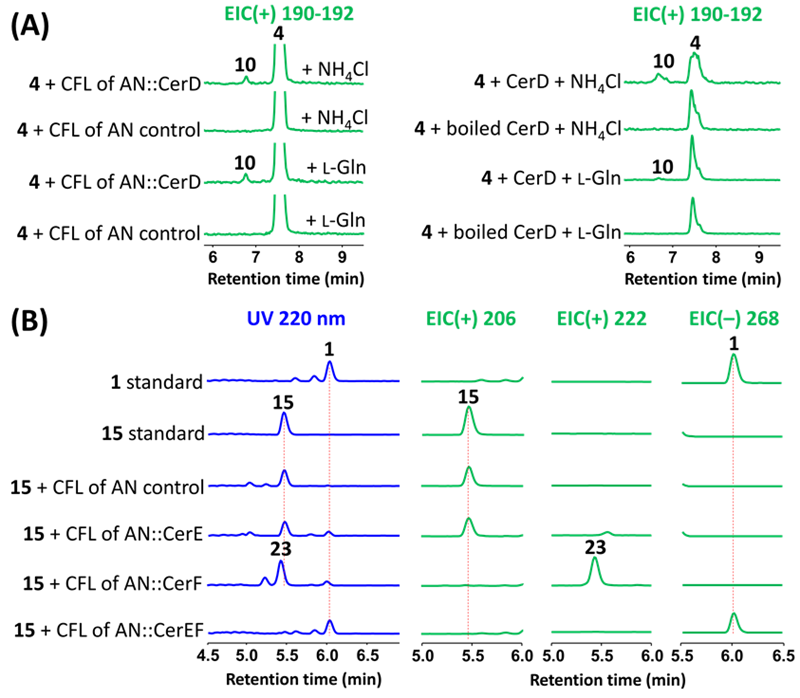
图5. 体外实验验证CerD、CerE和CerF的催化功能。图片来源:Angew. Chem. Int. Ed.
通过异源表达和体外实验,结合中间体和副产物的化学结构,作者提出了cerulenin(1)的生物合成途径:聚酮合酶CerA和硫酯酶CerB负责C12骨架的合成,形成具有2Z,4E,6Z,8Z,10Z构型的多不饱和脂肪酸中间体4,随后经过酰胺转移酶CerD的催化生成多烯酰胺10。10中的∆4双键被细胞色素P450单加氧酶CerP2与细胞色素b5 CerJ共同催化生成中间体(Ⅰ)。随后在CerC的催化下环氧开环、6Z,8Z双键迁移并异构化、10Z双键异构化,形成具有2Z,5E,7E,10E结构的中间体(II)。接下来,另一个细胞色素P450单加氧酶CerP1环氧化中间体(II)的∆2双键,生成关键中间体15。15中的4位羟基被FAD依赖的氧化酶CerF氧化成酮,再由CerE还原∆5双键,得到终产物cerulenin(1)。另外,作者也归纳了另外3条cerulenin生物合成过程中的旁路途径(shunt pathway)。
综上所述,作者利用自抗性基因导向的基因组挖掘、异源表达和XAD-16树脂吸附策略,发现并验证了两株Sarocladium真菌中负责cerulenin(1)生物合成的cer基因簇,并在构巢曲霉中实现了cerulenin合成途径的异源重构,首次揭示了cerulenin的生物合成途径。从具有E和Z混合构型的C12聚酮前体开始,涉及一系列复杂的酰胺化,环氧化,双键迁移,E/Z异构化和环氧开环等过程。通过体外反应,验证了氨基转移酶CerD、氧化酶CerF和还原酶CerE的功能。这是自60多年前发现FAS抑制剂以来,首次揭示cerulenin生物合成的分子基础,为cerulenin的工程化改造和放大生产提供了基础。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Self-Resistance Gene-Guided Discovery of the Molecular Basis for Biosynthesis of the Fatty Acid Synthase Inhibitor Cerulenin
Zhuo Shang*, Amr A Arishi, Changzheng Wu, Fangzheng Lao, Cameron L M Gilchrist, Stephen A Moggach, Ernest Lacey, Andrew M Piggott*, Yit-Heng Chooi*
Angew. Chem. Int. Ed., 2024, DOI: 10.1002/anie.202414941
尚卓教授简介

尚卓,山东大学教授,博士生导师,山东大学齐鲁青年学者(第一层次),2022年山东省海外优青获得者。2016年毕业于澳大利亚昆士兰大学,先后在加州大学圣地亚哥分校、洛克菲勒大学、南卡罗莱纳大学和西澳大学从事微生物活性天然产物的挖掘、生物合成和作用机制的研究工作。2022年6月加入山东大学药学院,依托“天然药物和海洋药物化学生物学交叉创新团队”(团队PI:鞠建华教授,国家杰青)开展研究工作。尚卓课题组主要从事微生物活性天然产物的研究,旨在通过生物信息学、分子生物学、天然产物化学、合成生物学和高通量筛选等多种交叉学科手段,挖掘新颖的微生物天然药源分子、解析其生物合成机制和作用机理,推动抗感染和抗肿瘤药物先导化合物的开发。迄今为止,以第一作者和通讯作者身份在Angew. Chem. Int. Ed.、J. Am. Chem. Soc.、Nat. Commun.等期刊发表研究论文42篇,承担国家级和省部级项目5项。担任Natural Product Reports、Organic Letters等13个国际同行评议学术期刊审稿人。详情可见个人主页:https://www.pharm.sdu.edu.cn/info/1058/13700.htm
因科研工作需要,尚卓课题组亟需招聘博士后和科研助理各一名,具体要求和待遇如下:
一、博士后招聘
【职位描述与要求】
1. 符合山东大学博士后招收的基本条件,博士毕业3年以内,有天然产物化学、微生物学、分子生物学专业背景者优先。
2. 具有较强的英语读写能力和团队协作精神,以第一作者身份在国际同行评议杂志上发表研究论文至少1篇。
3. 在合作导师的指导下,独立开展科研工作和科研项目的申报。
4. 能保证在山东大学药学院连续从事博士后研究工作不少于21个月。
【应聘方式】
申请人请将个人简历(包括个人学习、工作和研究经历)和代表性论文的pdf文件发送至 zshang@sdu.edu.cn,邮件标题注明“应聘-博士后-姓名”。应聘材料将予以严格保密,经初步审核通过者,课题组将尽快安排面试。其中特别资助类博士后申请以学校的申报要求为准。
二、科研助理招聘
【职位描述与要求】
1. 应聘者应具有硕士学位或即将获得硕士学位,从事天然产物化学、微生物分子遗传学或生物信息学等相关专业;会使用R或Python语言进行数据处理的应聘者优先考虑。
2. 能够在课题组长的指导下完成既定的科研工作,并定期向课题组长汇报工作进展。
3. 工作严谨,有责任心,有良好的沟通表达能力、团队协作意识和科研素养。
4. 能熟练使用Word和PowerPoint等办公软件,有较好的科研写作能力和英文文献阅读能力。
【应聘方式】
申请人请将个人简历(包括个人学习、工作和研究经历)及其他可以证明本人能力和学术水平的相关材料pdf文件发送至 zshang@sdu.edu.cn,邮件标题注明“应聘-科研助理-姓名”。应聘材料将予以严格保密,经初步审核通过者,课题组将尽快安排面试。
阅读详情,请使用下方二维码及链接

https://www.x-mol.com/news/902532
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!































 京公网安备 11010802027423号
京公网安备 11010802027423号