基于静态准分子聚集体自发闪烁的超快超分辨成像
单分子定位方法已被广泛用于获得生物结构的超分辨图像。然而,单个荧光团随机切换发光状态的低闪烁频率极大地限制了单分子定位显微镜(SMLM)的成像速度。近日,上海交通大学樊春海院士(点击查看介绍)、沈建磊副教授(点击查看介绍)团队报道了一种基于静态准分子聚集体自发闪烁的超快超分辨成像方法。
染料聚集体中的集体荧光闪烁是一种由荧光团之间的超快激发能量转移(EET)驱动的光物理现象。在染料聚集体中,被激发的荧光团可以将其能量转移到处于“ON”状态的分子上,从而产生荧光。相反,如果能量转移到淬灭分子,则荧光信号减弱,导致“OFF”状态。淬灭分子可以可逆地切换回荧光状态,导致发光状态和非发光状态之间的随机切换。这种超快EET诱导的集体荧光闪烁已在许多系统中观察到,包括共轭聚合物、树枝状聚合物、J-聚集体和染料掺杂的荧光纳米颗粒。然而,利用EET诱导的荧光闪烁进行超分辨成像的报道有限,其主要限制在于缺乏对染料聚集体的自组装行为的精确控制。
上海交通大学团队通过利用DNA框架结构上的花菁染料K21聚集体的自发荧光闪烁,开发了一种新型的超快SMLM技术。DNA模板可以作为“光捕获纳米天线”诱导静态准分子聚集体的形成,而静态准分子之间的EET导致染料聚集体的集体超快荧光闪烁。与传统的SMLM相比,这种基于静态准分子自发闪烁的策略使DNA纳米结构的成像速度提高了12.5倍。此外,该方法演示了在微流体系统中对DNA纳米结构进行20分钟以上的超分辨示踪。
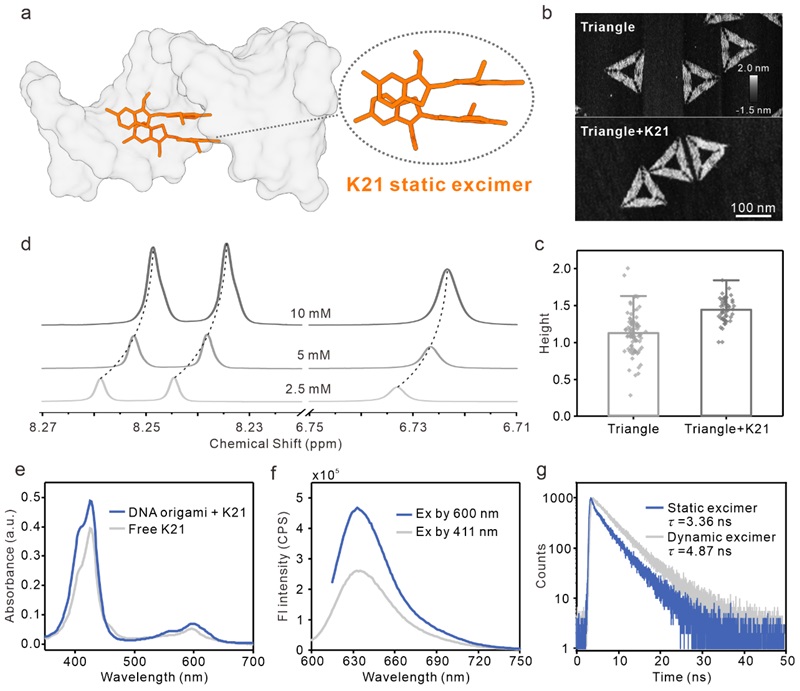
图1. DNA-K21静态准分子复合物的形成与表征
作者首先探究了K21在DNA纳米结构上的组装行为。通过1H NMR、吸收、荧光和寿命光谱,作者证明了K21可以以单体、动态准分子和静态准分子的形式在DNA模板上进行组装。而分子动力学模拟结果表明K21可以自发形成基态二聚体,即静态准分子,并嵌入DNA双螺旋的大沟中。

图2. DNA-K21静态准分子的荧光闪烁机制及动力学调控
之后,作者利用全内反射荧光(TIRF)显微镜对单个负载有K21静态准分子的DNA折纸进行了荧光动力学分析。与常规的探针SYTOX Orange和YOYO-1相比,静态准分子表现出更短的亮、暗时间,闪烁动力学分别加快了6.6和6.8倍。同时,作者利用FRET和荧光各向异性等实验方法证明了这种超快荧光闪烁源自于静态准分子之间的EET。并且,静态准分子的荧光闪烁动力学可以通过改变氧浓度、激光强度、染料负载密度和环境粘度等因素来调节。
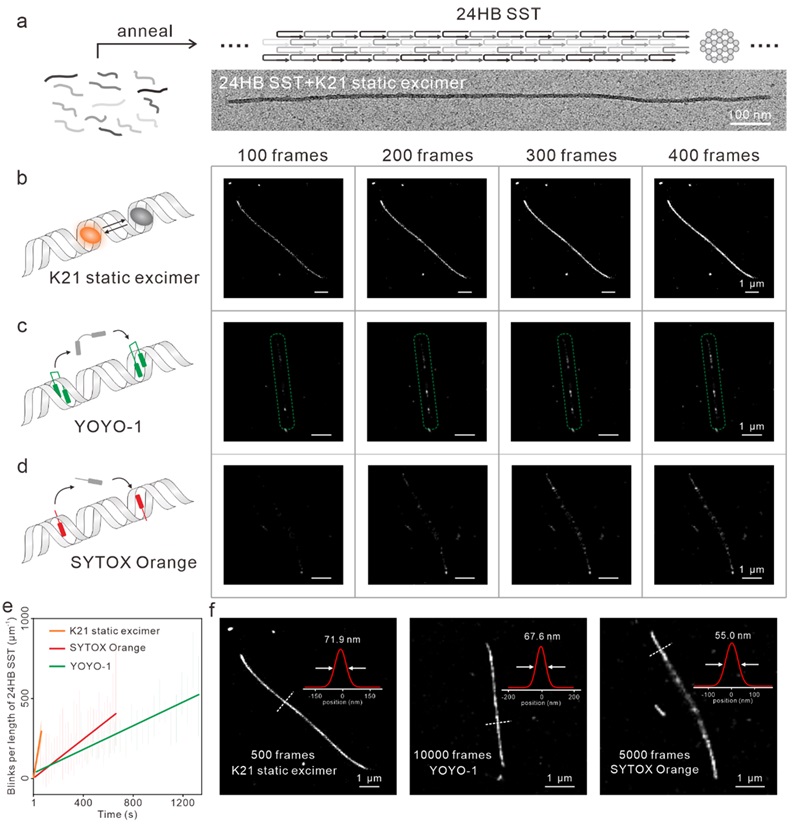
图3. 不同探针的超分辨成像速度的比较
随后,作者设计了一种24HB SST的长DNA结构,并利用K21静态准分子对其进行快速超分辨成像。在200帧(约27秒)内,静态准分子就使24HB的线性形貌变得清晰可见,而其他两种探针都无法在相同时间内重建出24HB的连续形貌。其中,静态准分子每秒的闪烁事件数比YOYO-1和SYTOX Orange分别高出约12.5和7.6倍。
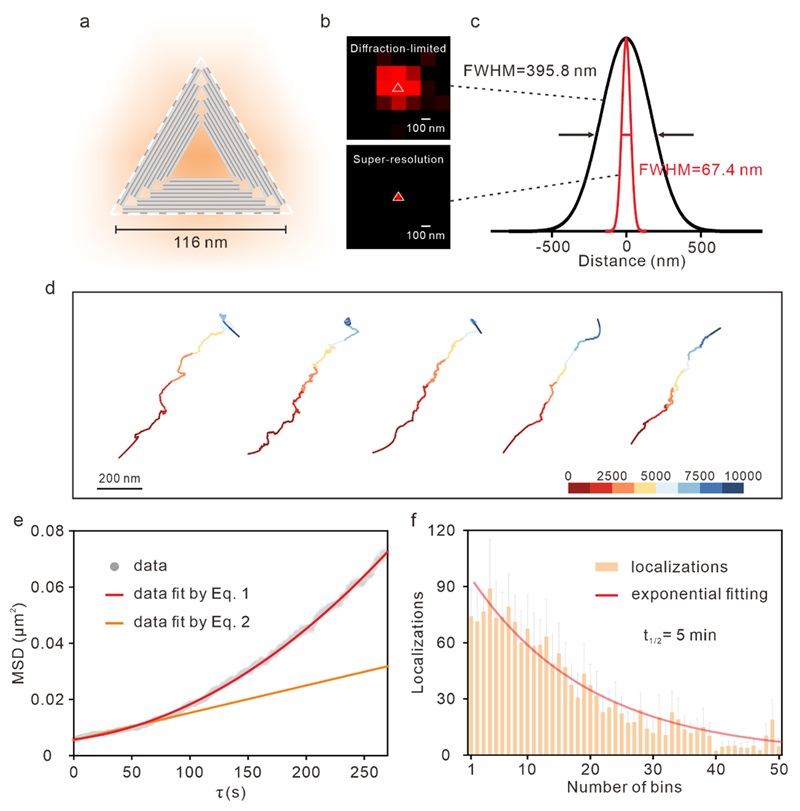
图4. 微流体系统中K21静态准分子对DNA纳米结构的长时间示踪
最后,利用K21静态准分子良好的光稳定性,作者成功实现了在微流体系统中对单颗粒DNA纳米结构的长时间超分辨示踪。
总之,DNA框架结构的使用为染料分子的可控组装提供了一条途径,以诱导其发生快速EET和荧光闪烁。该方法在DNA纳米结构的超分辨成像及长时间示踪等方面具有重要前景。
这一成果近期发表在J. Am. Chem. Soc.上,文章的第一作者是上海交通大学的博士生李聪和助理研究员谢晓冬。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Ultrafast Super-Resolution Imaging Exploiting Spontaneous Blinking of Static Excimer Aggregates
Cong Li, Xiaodong Xie, Mingqiang Li, Haozhi Wang, Xinyi Cheng, Jichao Zhang, Qian Li, Jiang Li, Xiaolei Zuo, Chunhai Fan*, and Jianlei Shen*
J. Am. Chem. Soc., 2024, 146, 18948–18957, DOI: 10.1021/jacs.4c01084
导师介绍
樊春海
https://www.x-mol.com/university/faculty/188467
沈建磊
https://www.x-mol.com/university/faculty/350886
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!

















































 京公网安备 11010802027423号
京公网安备 11010802027423号