可执行数学运算的核酸分子计算芯片用于癌症诊断
在机器学习领域,通过学习处理输入数据的分类算法能够产生更精确的分类结果。将这些分类算法应用于多组学数据的分析,可以显著提升医学诊断的精确度。例如,利用针对细胞外游离RNA的分类器可以预测先兆子痫的风险,而DNA甲基化图谱的分类器则可用于癌症的早期诊断和筛查。DNA分子遵循的沃森-克里克碱基配对规则赋予了它们可寻址和可编程的特性,这使得DNA可以被用来构建逻辑电路、神经网络等分子计算系统。同时,DNA作为关键的生物大分子,为生物分子识别和生物信息处理提供了天然的接口。目前,已有多种DNA分子分类器被开发出来,它们在图像识别、疾病早期诊断等领域展现出巨大的应用潜力。然而,现有的DNA分类器主要依赖于其可扩散分子组分间的相互作用,这在很大程度上限制了它们在错误校正能力以及构建大规模、高复杂度分子分类器方面的潜力。
为突破上述限制,中国科学院杭州医学研究所的韩达研究员团队开发了一种空间定位的DNA分类器用于癌症诊断,相关工作发表在Nature Communications 上。该工作将DNA分子计算系统以空间定位的形式整合在DNA折纸框架中构建DNA分类器(DNA integrated circuits-based classifier, DNA IC-CLA)以提高分类效率,且降低了设计复杂性。如图1所示,DNA IC-CLA以平面的矩形DNA折纸作为框架,以空间定位的信息分类运算模块为计算核心,通过toehold介导的DNA链置换反应(SDRs)对输入的核酸分子信号执行乘法、加法和减法运算,模拟了神经元对输入信号的加权、传递和输出。DNA IC-CLA通过对血清样本中的多个miRNAs输入执行数学运算,实现了对临床肺癌样本和健康样本的线性分类。与自由扩散的DNA分子电路相比,DNA IC-CLA能够在合成样本和临床样品中更快(约3 h)、更准确的诊断肺癌。
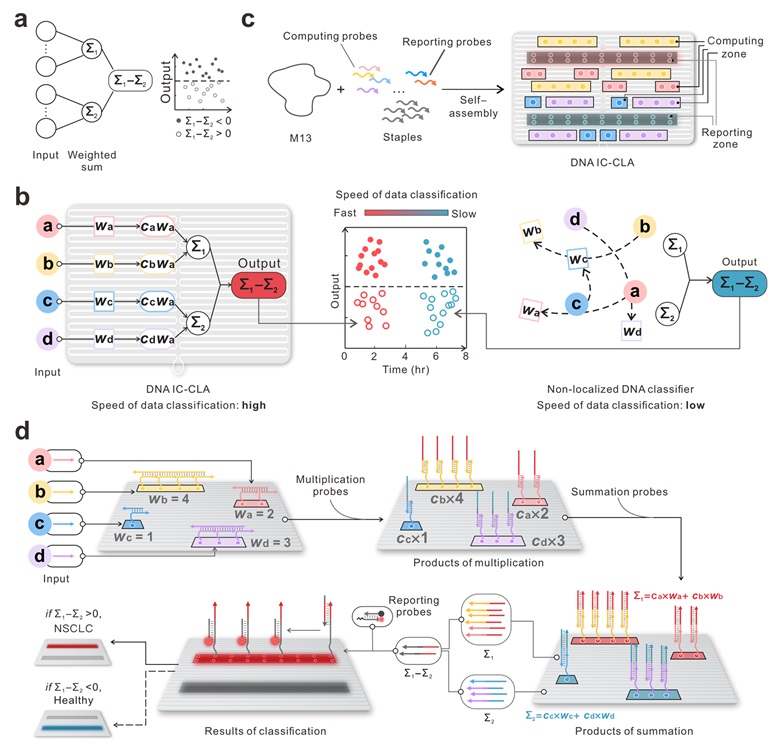
图1. DNA IC-CLA的构建及其应用于肺癌分子诊断示意图
对于给定的函数f(x) = 2×c(a) + 4×c(b) – 1×c(c) - 3×c(d),它包含了乘法、加法和减法三种运算。作者首先验证了在DNA IC-CLA上分别执行乘法、加法和减法的可行性。如图2a, b所示,DNA IC-CLA能够对输入的DNA信号a、b、c、d正确地执行f(x) = 2×c(a)、f(x) = 4×c(b)、f(x) = 1×c(c)、f(x) = 3×c(d),即给指定的输入赋予权重。进一步地,也能将加权值进行加和运算,即f(x) = 2×c(a) + 4×c(b)、f(x) = 1×c(c) + 3×c(d)并报告对应强度的荧光信号 (图2c, d)。诊断结果的给出需要通过减法运算对加权求和值进行比较。作者以f(x) = 2×c(a) + 4×c(b) 的输出信号E和f(x) = 1×c(c) + 3×c(d) 的输出信号F为输入验证了它们在不同化学计量比下的减法运算的正确性 (图2e, f, g)。上述结果显示DNA IC-CLA可以正确地执行乘法、加法和减法运算,即能对输入的核酸信号进行加权、加权求和以及加权求和值大小比较。
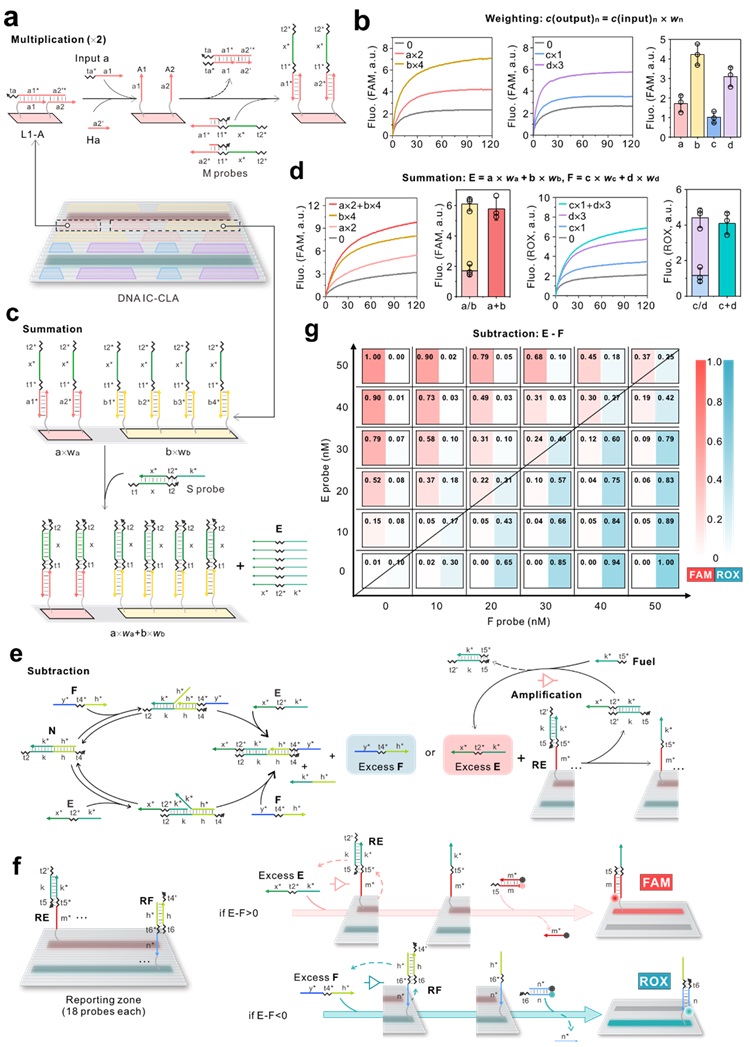
图2. DNA IC-CLA的运算模块设计及用于乘法、加法和减法运算的验证
如图3a所示,相较于自由扩散的分子运算系统,DNA IC-CLA在包含3步SDRs的乘法运算和包含4步SDRs的加法运算中都表现出更快运算速度,其反应速率比自由扩散系统快1-1.5倍。在超过5步SDRs的减法运算中,DNA IC-CLA的反应速率达到了自由扩散系统的3倍。此外,作者比较了两种计算系统完成一个完整的函数运算分别所需的时间,该函数f(x) = 2×c(a) + 4×c(b) – 1×c(c) - 3×c(d) 包含6步SDRs和4种不同的DNA输入。DNA IC-CLA在1.3小时内完成计算,而自由扩散系统则需要超过3小时,两种系统的反应速率差异超过3倍。这些结果证实空间定位的DNA分子计算模块可以显著提高系统计算速度。此外,这种模块化设计的系统还显示出更高的稳健性,DNA IC-CLA在组装1周后仍然可以准确执行有效的算术运算,而自由扩散系统在制备、混合1周后表现出变低的信噪比 (图3b)。这可能是因为DNA组分在DNA折纸框架上的空间定位减少了组分间的窜扰和自发的分子间相互作用。且作者在此验证了DNA IC-CLA能够正确地执行f(x) = 2×c(a) + 4×c(b) – 1×c(c) - 3×c(d),给出与理论计算结果相对应的荧光信号 (图3c)。总的来说,DNA IC-CLA在执行分类任务时比自由扩散的系统更快、更稳定。
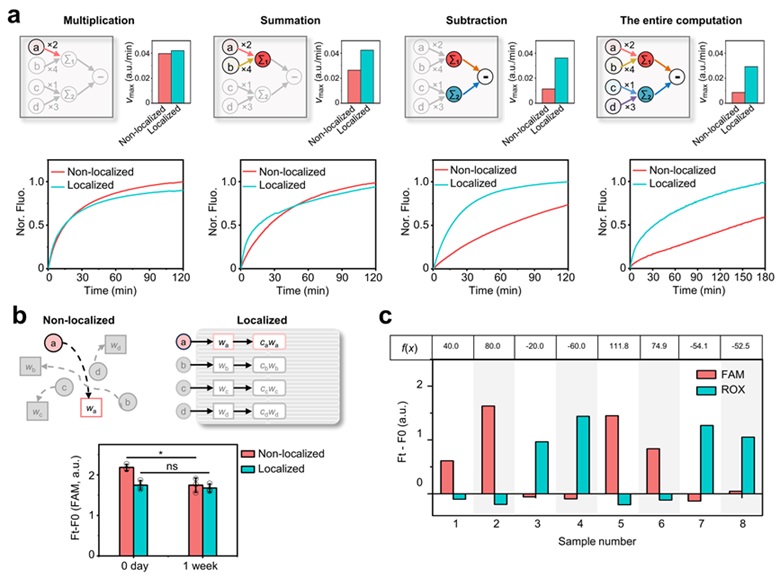
图3. DNA IC-CLA与自由扩散的DNA系统的性能比较
在该工作的最后部分,作者将DNA IC-CLA应用于合成的miRNAs样本和临床血清样本的,验证其对非小细胞肺癌和健康样本的区分效果。如图4a-d所示,DNA IC-CLA对合成样本检测的准确率达到90.0%,证实DNA IC-CLA可以像人工神经元一样对输入的核酸分子信号进行加权、加和以及信息传导,实现合成miRNAs样本的正确分类。将DNA IC-CLA应用于临床血清样本时 (图4e, f),尽管相较于合成miRNAs样本,DNA IC-CLA对临床样本的分类准确率下降,但也达到78.0%的检测准确率,这一结果证明DNA IC-CLA能够识别、区分多个生物标记物并在不需要人为干预情况下实现肺癌诊断。
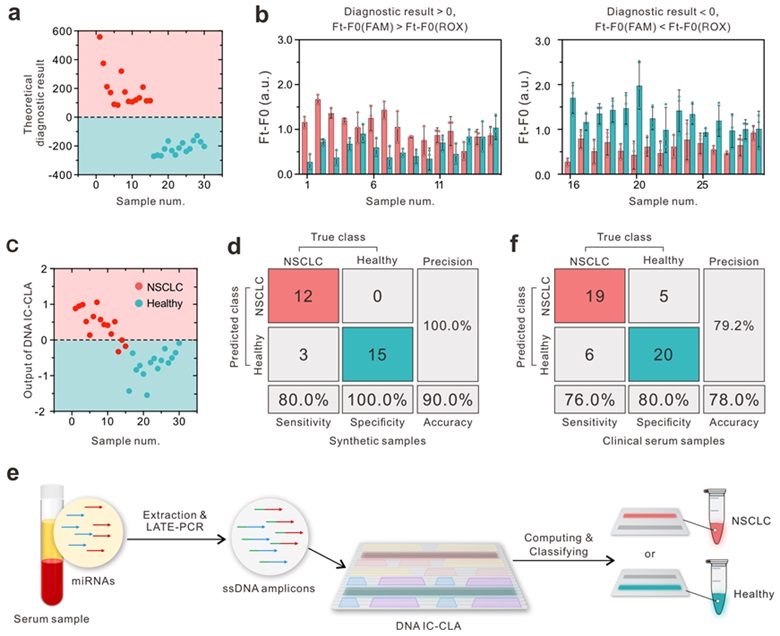
图4. DNA IC-CLA对合成样本和临床样本的分类结果
小结
本研究开发了一种基于类脑运算、空间定位的DNA分子计算芯片,通过将DNA分子计算核心集成到DNA折纸框架的表面用于分析多生物标记物输入,实现癌症诊断。加速决策过程和提高诊断精度对于临床应用至关重要。DNA折纸框架提供的空间约束有助于加速DNA分子计算的动力学,并减少不同区域特别是相距较远的探针之间的窜扰。通过提高DNA IC-CLA的分类速度显著了缩短诊断时间,避免运算模块之间的窜扰降低了错误诊断的概率。相信在未来DNA IC-CLA这种一体化的核酸分子芯片能够作为微、纳尺度上的分子芯片的一砖一瓦,构筑更大规模、具备更高阶功能的智能分子体系,并在细胞生物计算和分子医学诊断中展示更多的应用。
该工作得到国家重点研发计划、国家自然科学基金等项目支持,杨林林博士和唐倩博士为该论文的共同第一作者。韩达研究员、张朝副研究员和郭沛副研究员为通讯作者。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

A spatially localized DNA linear classifier for cancer diagnosis
Linlin Yang, Qian Tang, Mingzhi Zhang, Yuan Tian, Xiaoxing Chen, Rui Xu, Qian Ma, Pei Guo, Chao Zhang & Da Han
Nat. Commun., 2024, 15, 4583, DOI: 10.1038/s41467-024-48869-y
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!
































 京公网安备 11010802027423号
京公网安备 11010802027423号