南方科技大学Nat. Commun.:机器学习赋能加快多尺度量化精修可靠的蛋白质–药物结构
生物大分子的精确三维结构在药物开发和生物催化中至关重要。X-射线衍射 (XRD) 是解析生物大分子三维原子结构的最常用的方法之一,科学家通常采用X-射线晶体精修方法,结合分子力学 (MM) 力场与实验 (XRD) 数据来解析生物大分子的三维结构并构建合理的化学结构。另一方面,Ryde等人提出了量化精修 (quantum refinement) 方法,将更可靠的量化计算 (QM) 方法替代快速的分子力学力场描述蛋白活性中心,能够有效改善关键局部结构 (J. Comput. Chem. 2002, 23, 1058) 。此外,其他课题组也将量化精修与多尺度模拟、线性标度QM、分块法等方法相结合以进一步加快或改进精修过程。2021年,南方科技大学钟龙华(点击查看介绍)课题组提出了基于ONIOM多尺度量化计算方法的量化精修方法 (ONIOM_QR) ,并成功提高了金属蛋白活性中心的关键局部结构精确度 (J. Chem. Theory Comput. 2021, 17, 3783) ,但可靠的多尺度QM/MM、QM/SE或QM/SE/MM方法计算比较耗时(特别采用高水平的量子嵌入CCSD-in-DFT方法作为QM方法),阻碍了量化精修在生物体系中的广泛应用。

图1. 研究方法示意图
最近,该课题组进一步结合了高效的机器学习势 (MLPs) 和多尺度量化精修方法 (ONIOM_QR) ,以提高量化精修的效率和准确性(图1和图2a)。首先,他们从PDBbind v2020 数据集中建立含有12,963个药物/抑制剂分子的计算测试集 (PB20-QM)和两个子计算测试集(含C, H, O, N, F, S和Cl 元素为主的PB20-QM-8K测试集和含C, H, O和N元素为主的PB20-QM-3K测试集),并采用比较可靠的DFT方法 (ωB97X-D) 与几个通用型的机器学习势 (MLPs) 和半经验 (SE) GFN2-xTB方法对比计算结构的准确性(图2c)。为了突破MLPs对于元素类别的限制,他们首次通过ONIOM策略将不同精度 (耦合簇 (CC) 和DFT精度) 的MLPs结合起来(例如ONIOM2(ANI-1ccx:ANI-2x),ONIOM2(AIQM1:ANI-2x),或通过和半经验方法结合 (ONIOM2(MLP:SE)) 将MLP作为ONIOM方法中的最高水平计算方法来描述药物分子的关键中心结构(图2b),从而将MLPs扩展应用到更广泛的范围。然后,他们成功地对50种不同的蛋白质-药物/抑制剂体系进行了多尺度量化精修,从密度差值标准分数(RSZD scores)、形变能、键长、键角、转动二面角等方面验证了机器学习势 (MLPs) 在量化精修中准确性(图2d)。相对于ANI系列方法,耦合簇精度的AIQM1表现出更高的精度(特别对带电体系)。
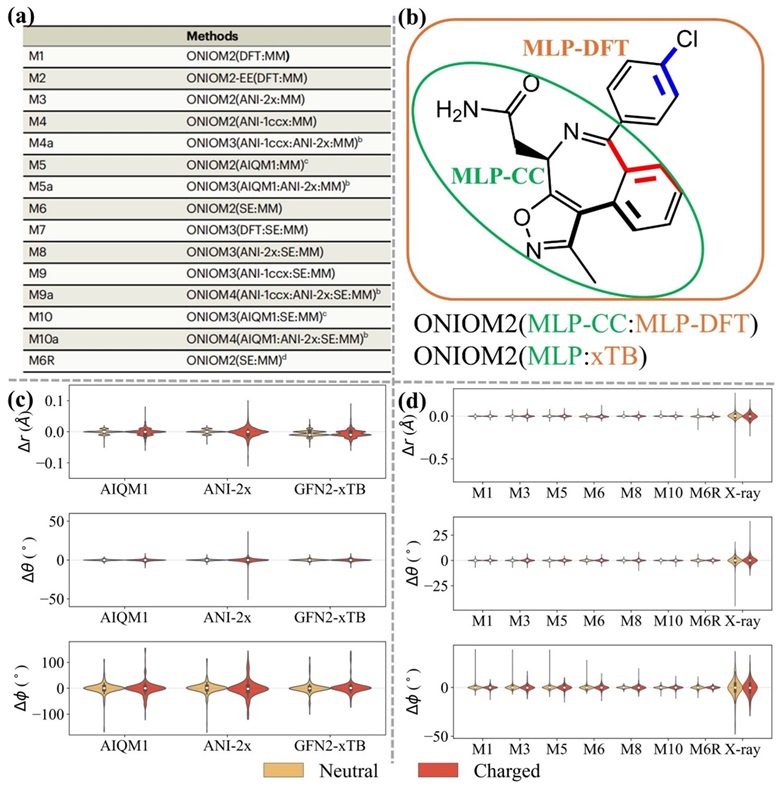
图2. (a) 不同组合的多尺度量化精修方法。(b) ONIOM策略结合不同精度的MLPs和xTB示意图。50个药物/抑制剂体系不同方法在(c)气相优化,(d)蛋白量化精修中键长、键角、旋转二面角结果对比。
此外,在对一个SARS-CoV-2主蛋白酶-Nirmatrelvir (FDA批准的新冠药物 Paxlovid) 晶体结构 (PDB ID: 7RFW) 进行量化精修时,发现Nirmatrelvir与Cys145存在成键与非成键形式共存构象的情况,通过量化精修估计了这两个构象的可能占比(约为7:3)并大大提高了该晶体药物局部结构精度(图3),期望该量化精修结果为新冠药物的设计研发提供新的结构视角。
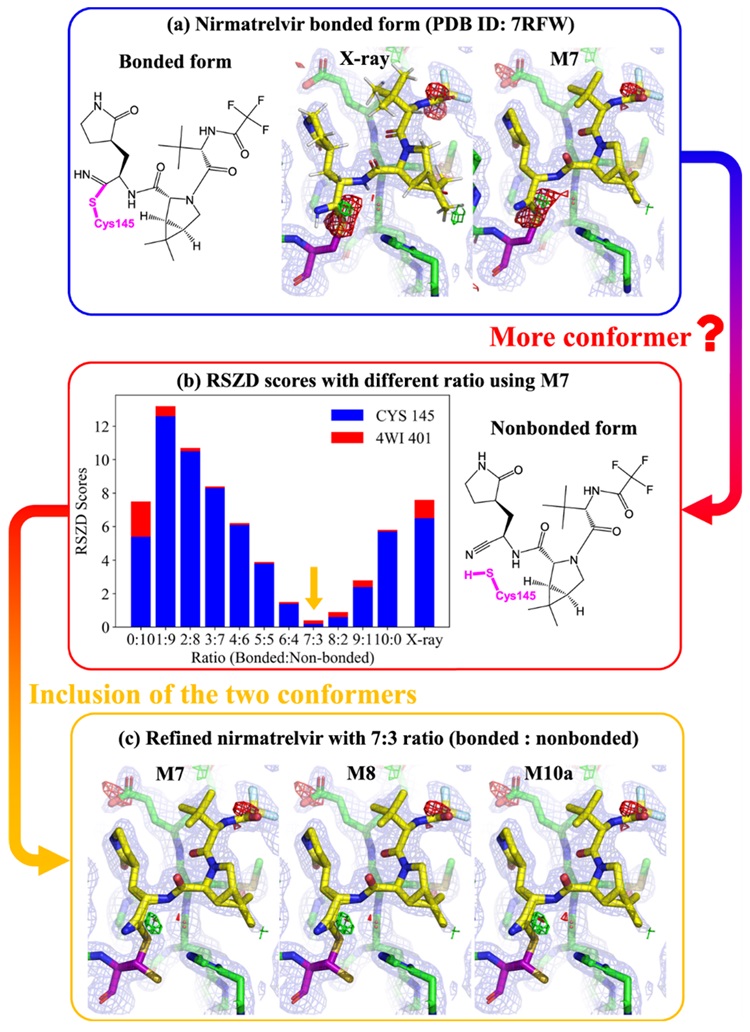
图3. Nirmatrelvir 在SARS-CoV-2主蛋白酶中与Cys145的成键形式构象 (a) 与非成键形式构象 (b),以及量化精修后结合两种构象不同占比的密度差值标准分数,(c) 不同方法量化精修与加入两种构象后的电子密度图。
该研究期望机器学习势 (MLPs) 能够应用在更加广泛和新型的生物分子结构实验解析方法,如冷冻电镜和微晶电子衍射 (Cryo-EM,MicroED) 。同时,新的药物分子计算数据库 (PB20-QM、 PB20-QM-8k和PB20-QM-3k) 期望帮助推动发展更好的DFT、MLPs和SE方法,为广泛药物分子提供可靠的结构预测。
这一成果近期发表在Nature Communications上,文章的第一作者为南方科技大学的高级研究学者严泽印,通讯作者是南方科技大学的钟龙华教授。南方科技大学的硕士生韦大聪和李欣教授也对该工作做出了贡献。该研究得到了国家自然科学基金、广东省催化重点实验室和深圳市自然科学基金的支持。部分计算资源得到了南方科技大学计算科学与工程中心和化学系高性能超级计算集群的支持。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Accelerating reliable multiscale quantum refinement of protein–drug systems enabled by machine learning
Zeyin Yan, Dacong Wei, Xin Li & Lung Wa Chung*
Nat. Commun., 2024, 15, 4181. DOI: 10.1038/s41467-024-48453-4
导师介绍
钟龙华
https://www.x-mol.com/university/faculty/39474
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!


































 京公网安备 11010802027423号
京公网安备 11010802027423号