贺福初、李栋团队发布首个人类泛素连接酶-底物相互关系预测系统丨亮点推荐
本文获得“BioArt”授权转载
BioArt按:泛素-蛋白酶体途径是真核生物最重要的蛋白质降解径。根据蛋白泛素位点数据库mUbiSiDa统计,目前有超过5700个底物蛋白的约30000个泛素位点被收录,然而只有一少部分(~15%)的底物蛋白有已知对应的E3泛素连接酶信息。近日,国家蛋白质科学中心(凤凰中心)贺福初院士、李栋研究员团队在Nature Communications杂志上发表了题为“An integrated bioinformatics platform for investigating the human E3 ubiquitin ligasesubstrate interaction network”的论文,对泛素连接酶与底物的相互作用涉及的蛋白质网络、结构和序列等多个层面的生物大数据开展了系统分析,给出了3856对潜在的介导泛素连接酶与底物相互作用的结构域组合,鉴定了底物蛋白序列上10480个潜在的泛素连接酶识别特征(motif),发现了泛素连接酶与底物在生物学网络中倾向于形成特定的拓扑性质,并且基于这些特征研发了首个人类泛素连接酶-底物相互作用的预测和展示系统UbiBrowser。该项研究有助于人们从多个角度认识泛素连接酶对底物的调控作用,发现两者之间的选择关系,并进而掌握开启蛋白质死亡之门的“钥匙”。

论文解读:
蛋白质是自然界中最神秘的物质之一,几乎所有的生命活动都有它的身影。正如人有生死,生物体内的蛋白质也有出生与死亡。蛋白质的出生是一个精准的从脱氧核糖核酸 (DNA)转录得到信使核糖核酸(mRNA)进而翻译合成蛋白质的过程。当蛋白质的功能使命完成后,需要被及时降解掉(死亡),否则,蛋白质过早或者过晚降解均会造成其功能失常,进而导致“阿尔兹海默综合征”、“克雅士病”等多种疾病的产生。
泛素-蛋白酶体途径是真核生物最重要的蛋白质降解途径。在这个途径中,蛋白质被特异进行泛素标记(即泛素化修饰),进而被送到蛋白酶体进行降解。该途径是一种能量(ATP)依赖的特异、高效的蛋白质降解途径, 发现该途径的三位科学家Aaron Ciechanover、Avram Hershko与Irwin Rose共享了2004年诺贝尔化学奖。在该途径中,泛素连接酶发挥了“扳机”似的重要作用,特异地进行底物蛋白质的识别,并启动后续的降解过程,是打开蛋白质死亡之门的钥匙。
近年来,生物大数据的积累使得我们有望利用知识挖掘思想对这一问题进行理论探索。国家蛋白质科学中心贺福初院士、李栋研究员团队对泛素连接酶与底物的相互作用涉及的蛋白质网络、结构和序列等多个层面的生物大数据开展了系统分析,给出了3856对潜在的介导泛素连接酶与底物相互作用的结构域组合,鉴定了底物蛋白序列上10480个潜在的泛素连接酶识别特征(motif),发现了泛素连接酶与底物在生物学网络中倾向于形成特定的拓扑性质。进而基于这些特征研发了首个人类泛素连接酶-底物相互作用的预测和展示系统(UbiBrowser,http://ubibrowser.ncpsb.org)。
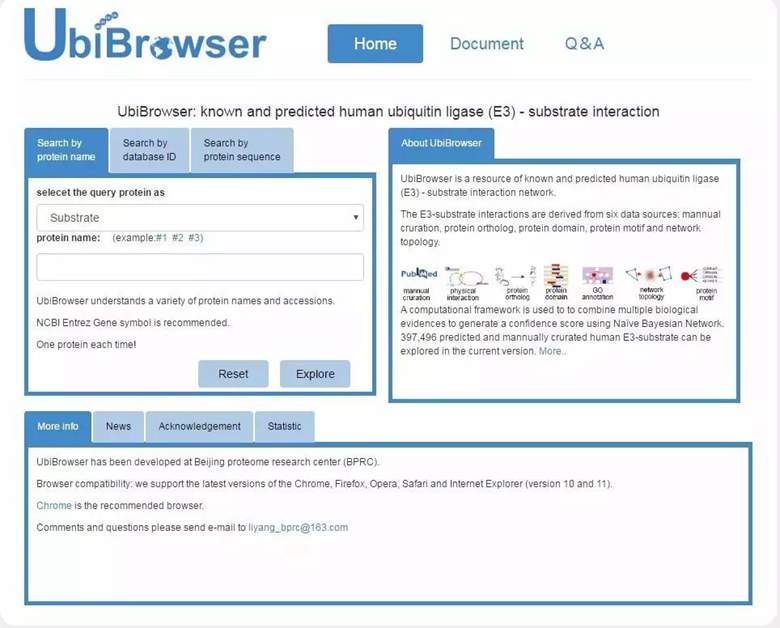
UbiBrowser支持多种数据提交方式,利用网络视图、列表视图和序列视图,展示了预测的泛素连接酶-底物相互作用、文献来源的泛素连接酶-底物相互作用以及蛋白的泛素化修饰位点和可能的被识别结构域和motif信息。模型预测有关的支持证据信息也同时向用户进行展示。该项研究有助于人们从多个角度认识泛素连接酶对底物的调控作用,发现两者之间的选择关系,并进而掌握开启蛋白质死亡之门的“钥匙”。
本项研究也是贺福初院士、李栋研究员团队利用生物大数据挖掘生物学知识的再次成功尝试。2008年,该团队整合基因组上下文等生物学特征,成功进行了蛋白质相互作用可靠性评估(Mol Cell Proteomics, 2008, 7: 1043-1052)。2009年,该团队利用结构域预测了蛋白质组尺度内相互作用蛋白间的信号流走向(Mol Cell Proteomics, 2009, 8: 2063-70)。2013年,该团队利用logistic回归方法,整合蛋白质功能等多层次生物学证据,对蛋白质组尺度的自相互作用蛋白进行了系统挖掘 (Mol Cell Proteomics, 2013,12:1689-700)。近年来,在国家科技部“中国人类蛋白质组计划”重点专项项目的支持下,该团队致力于构建面向生物医学大数据知识智能发现的“高速公路”。
据悉,该研究第一作者是国家蛋白质科学中心李杨博士、卢亮博士和首都医科大学谢萍副教授。贺福初院士和李栋研究员为共同通讯作者。该工作得到国家国际科技合作专项、精准医学重点专项和国家自然科学基金的支持。
参考文献:
Li, D., Liu, W., Liu, Z., Wang, J., Liu, Q., Zhu, Y., & He, F. (2008). PRINCESS, a protein interaction confidence evaluation system with multiple data sources. Molecular & Cellular Proteomics, 7(6), 1043-1052.
Liu, W., Li, D., Wang, J., Xie, H., Zhu, Y., & He, F. (2009). Proteome-wide prediction of signal flow direction in protein interaction networks based on interacting domains. Molecular & cellular proteomics, 8(9), 2063-2070.
Liu, Z., Guo, F., Zhang, J., Wang, J., Lu, L., Li, D., & He, F. (2013). Proteome-wide prediction of self-interacting proteins based on multiple properties. Molecular & Cellular Proteomics, 12(6), 1689-1700.

贺福初,蛋白质组学家,中科院院士。1982年毕业于复旦大学生物系,现任军事医学科学院研究员,是人类蛋白质组计划国际执委会成员、亚太蛋白质组组织主席。他是我国蛋白质组学的主要奠基者与学术带头人,在国内率先引进并建立蛋白质组学支撑技术平台,相继开展胎肝、成人肝、肝癌、细胞分化、凋亡、SARS等相关蛋白质组研究,为重大疾病的防诊治提供了新思路新策略。倡导并领衔了人类第一个组织、器官的“肝脏蛋白质组计划”,领衔实施“中国人类蛋白质组计划”。率先提出人类蛋白质组计划的科学目标与技术路线,领导揭示了人体首个器官-肝脏的蛋白质组及其连锁图。曾获国家级奖励6项,省部级一等奖8项。

李栋,军事医学科学院放射与辐射医学研究所、北京蛋白质组研究中心研究员,硕士生导师。北京市优秀青年人才,总后勤部优秀青年科技人才扶持对象,Proteomics编委。主要从事蛋白质组生物信息学研究。承担国家国际科技合作专项项目和国家自然科学基金等各类项目9项,首次整合多生物学证据进行高通量蛋白质相互作用数据的可靠性分析;建立国际上首个基于蛋白质网络预测信号通路的生物信息算法系统;首次揭示蛋白质相互作用网络拓扑上的“保守直径”现象和为进化上的“蛋白质成簇添加”现象提供模体层次的证据; 建立大规模组学数据可视化和分析软件平台,为国内外提供数据分析服务25000多次;对最大规模组织层次蛋白质网络——肝脏蛋白质相互作用网络进行研究。在Nature Communications、Molecular&Cellular Proteomics, Molecular Systems Biology和J Proteome Res等期刊发表第一/通讯作者(含并列)署名SCI论文10多篇,获国家科技进步创新团队奖等国家和省部级奖励5次,获软件著作权7项。
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!


































 京公网安备 11010802027423号
京公网安备 11010802027423号