Nature封面:像真正“折纸”的DNA折纸
2006年,美国科学家Paul Rothemund发明了一项名为“DNA折纸(DNA origami)”的技术,用一条具有7000个碱基对的DNA链段,通过弯曲、折叠出一个笑脸 [1]。这项工作被誉为DNA纳米技术领域的一项里程碑,极大促进了纳米尺度DNA结构的精确构建。近年来,科学家借鉴该技术,从1维到3维,从对称性到非对称性,完成了多尺度空间复杂结构的自组装,更推进了DNA分子计算机、DNA分子机器等多领域的发展。

DNA折纸术(第7082期Nature杂志封面)。图片来源:Nature [1]
有意思的是,“DNA折纸”这个名称,很可能是个美丽的误会。樊春海院士曾在一次报告中评价,Paul Rothemund的工作“更像我们织毛衣的过程”——DNA就像很柔软的一条毛线,当几百条短链碰撞在一起时,就像织毛衣一样,编织成所要的形状。“DNA折纸”这个名字,某种程度上可能是个错误。他不了解织毛衣,认为这更像是折纸,就把这项技术命名为DNA折纸。这是一个“错误”的开始,但是它带来了美丽,带来了整个领域的繁荣。[2,3]
近日,韩国首尔大学Do-Nyun Kim课题组基于折纸机理(paper-folding mechanism),实现了可编程、可重构的DNA折纸(DNA origami)结构。他们借鉴儿童折纸中一张纸可重复折叠和展开成不同形状的思路,设计了DNA线框纸(DNA wireframe paper)结构(下图a),可以将其折叠成各种目标形状,并实现了可编程、可重构的DNA折纸,并讨论了折叠动力学及技术的可扩展性。该工作在Nature 杂志上发表,并被选为封面文章。
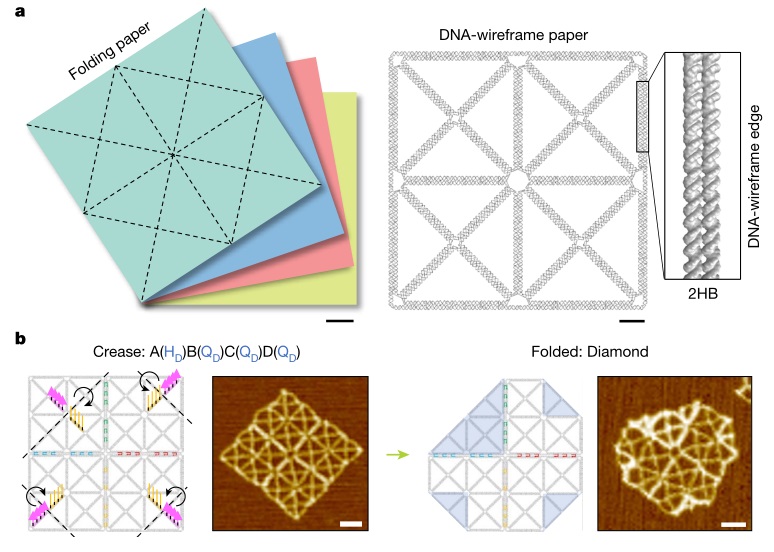
儿童折纸与本文DNA折纸。图片来源:Nature [4]

当期封面。图片来源:Nature
研究者设计的这种“DNA线框纸”,由DNA螺旋束充当中间的“折痕线”(根据需要设计成某种几何图案)以及“边缘”(刚性更高以保持整体结构的完整性),可沿着折痕线折叠和展开“纸”的某些部分。“折痕线”边缘有两类单链DNA作为“折痕手柄(crease handle)”,用于“纸”的折叠和展开。其中,通过加入与两类“折痕手柄”序列互补的“胶水链”,实现折叠过程;“胶水链”末端都有一个5 nt长的toehold,当加入与“胶水链”互补的“释放链”时,可以通过toehold介导的DNA链置换来展开已经折叠的“纸”结构(下图c)。
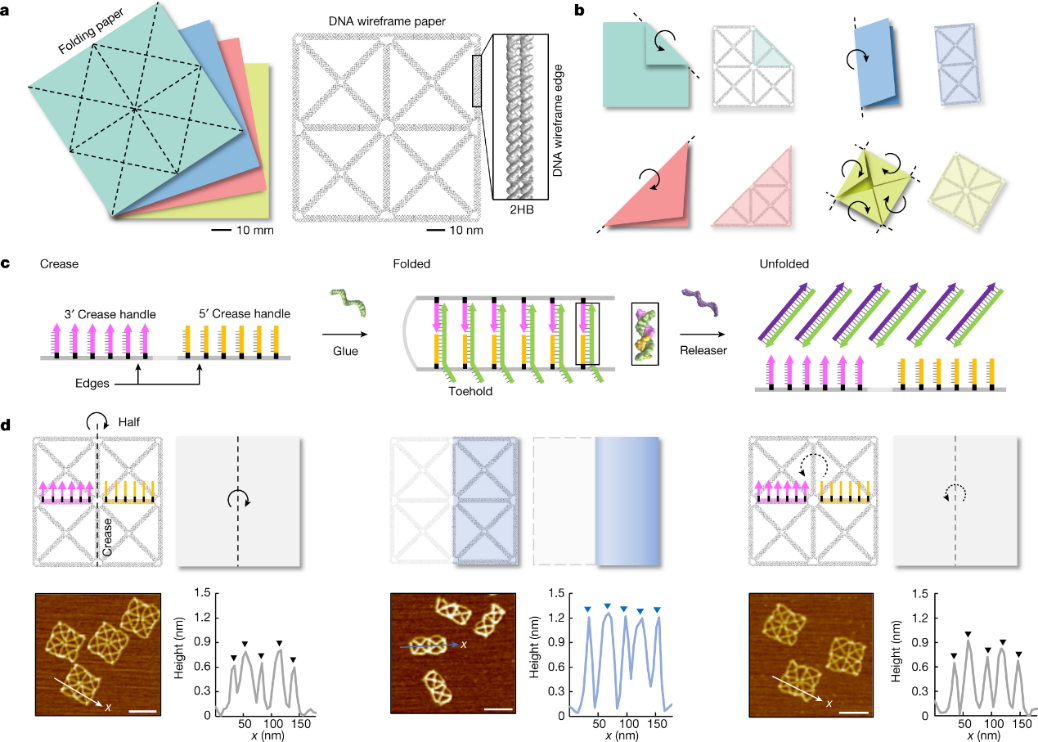
DNA折叠机理。图片来源:Nature
该方法重要的优点在于,可以操控“DNA线框纸”按照可编程的方式折叠成各种目标形状。研究者构建了8种折叠图案,AFM测量证实,所有8种图案都可以按预期设计成功折叠。
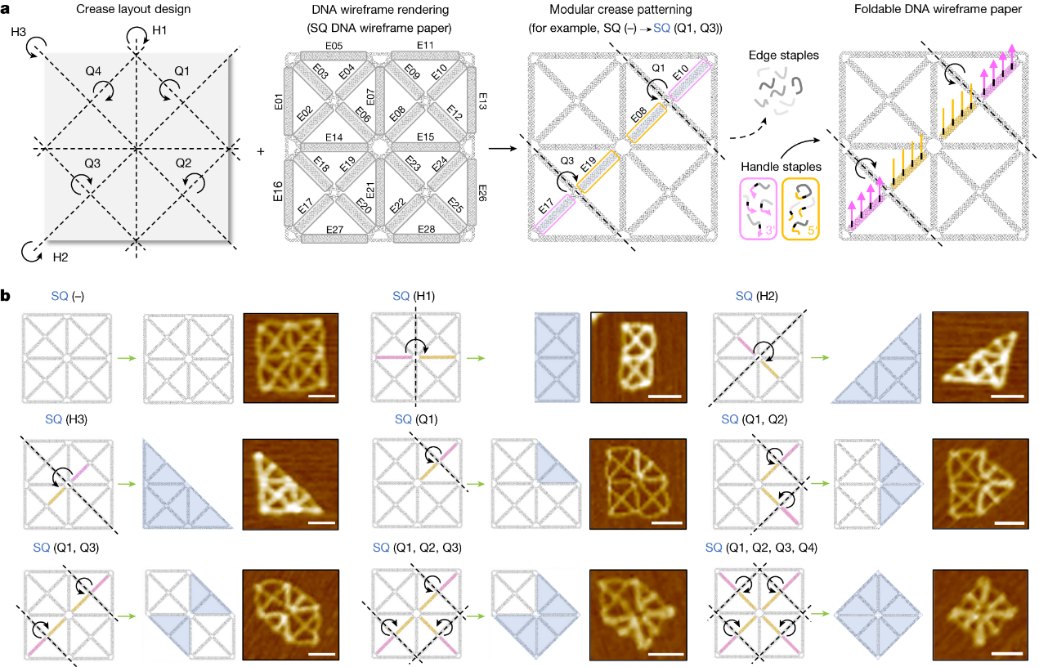
可编程的各种DNA折叠图案。图片来源:Nature
和我们小时候玩的折纸一样,折痕顶点过于坚硬,或者胶水不够强力,都可能会造成折叠图案的失败。为此,研究者优化了折痕设计和顶点刚度,通过增加“折痕手柄”数量以及调节柔韧性,最大限度地提高折叠成功率,最终使H1、H2和Q1三种折叠的产率从68%、38%和73%,分别提高至93%、94%和88%。
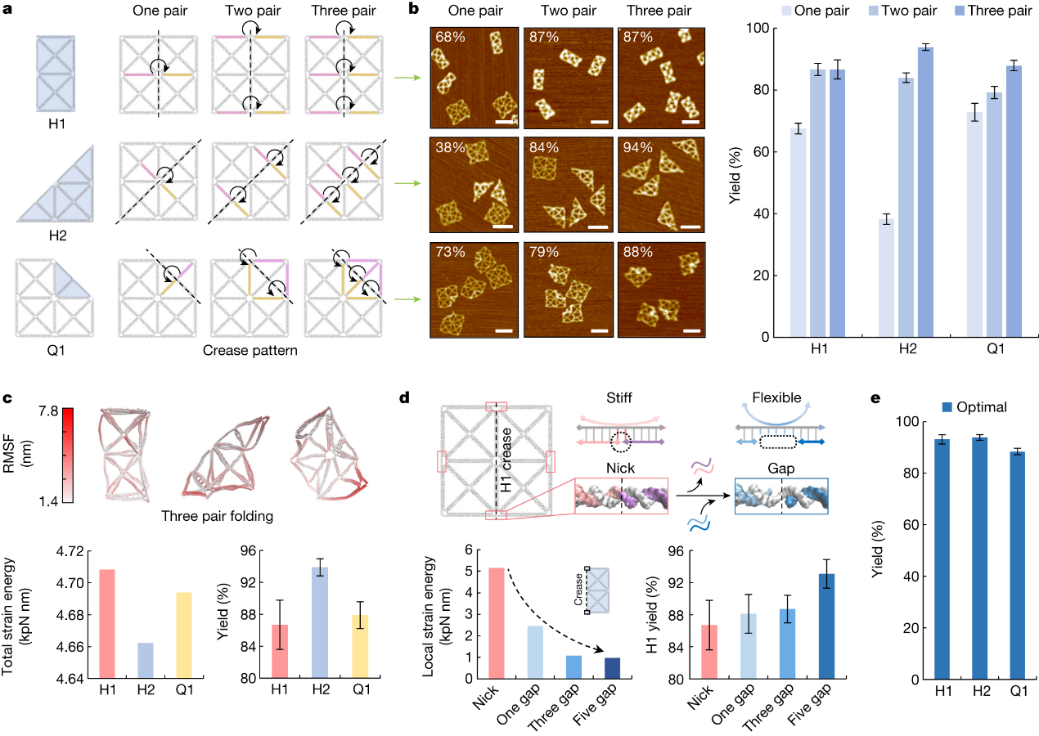
折叠产率优化。图片来源:Nature
这种折纸方式具有良好的可逆性,DNA线框纸可重复折叠和展开。向折叠后的溶液中加入“释放链”后,几乎立即完成展开反应。

折叠特性。图片来源:Nature
此外,这种可重复的折叠和展开不仅可以通过核酸链的加入来实现,还可以通过环境刺激来实现,例如pH值的变化、紫外线照射等。这将有助于提高可折叠DNA线框纸的尺寸,扩展其应用范围。
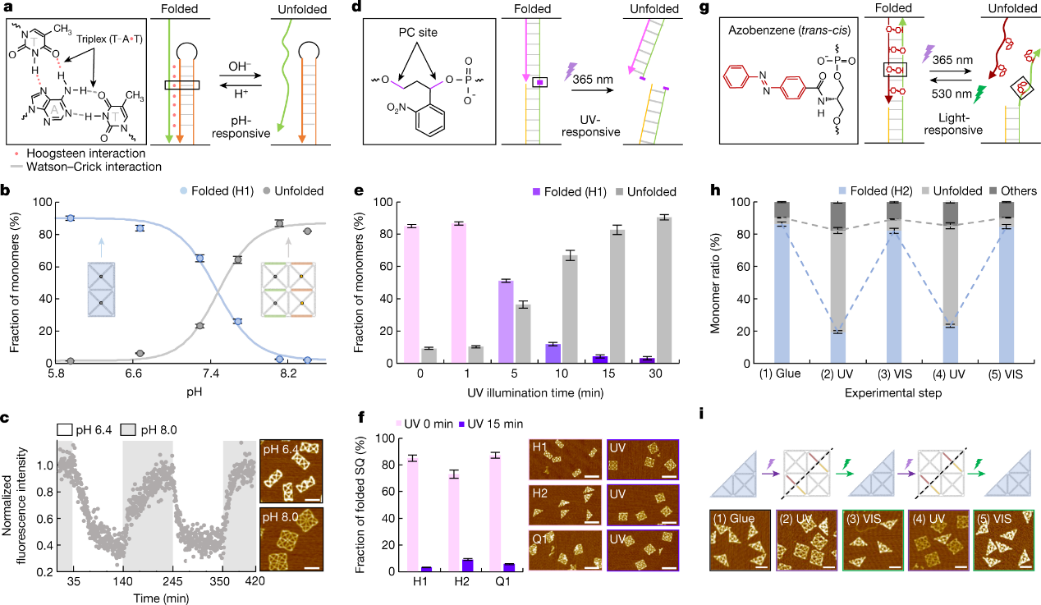
通过pH和光照控制折叠。图片来源:Nature
随后,研究者将DNA线框纸扩大了四倍,完成了4 × 4折叠矩阵编码。考虑到旋转对称性,一共有35种折叠情况。有趣的是,有些折叠方式还具有协同效应,即一种折叠有助于另一种折叠,可以使得两种设计的折叠产率提高。
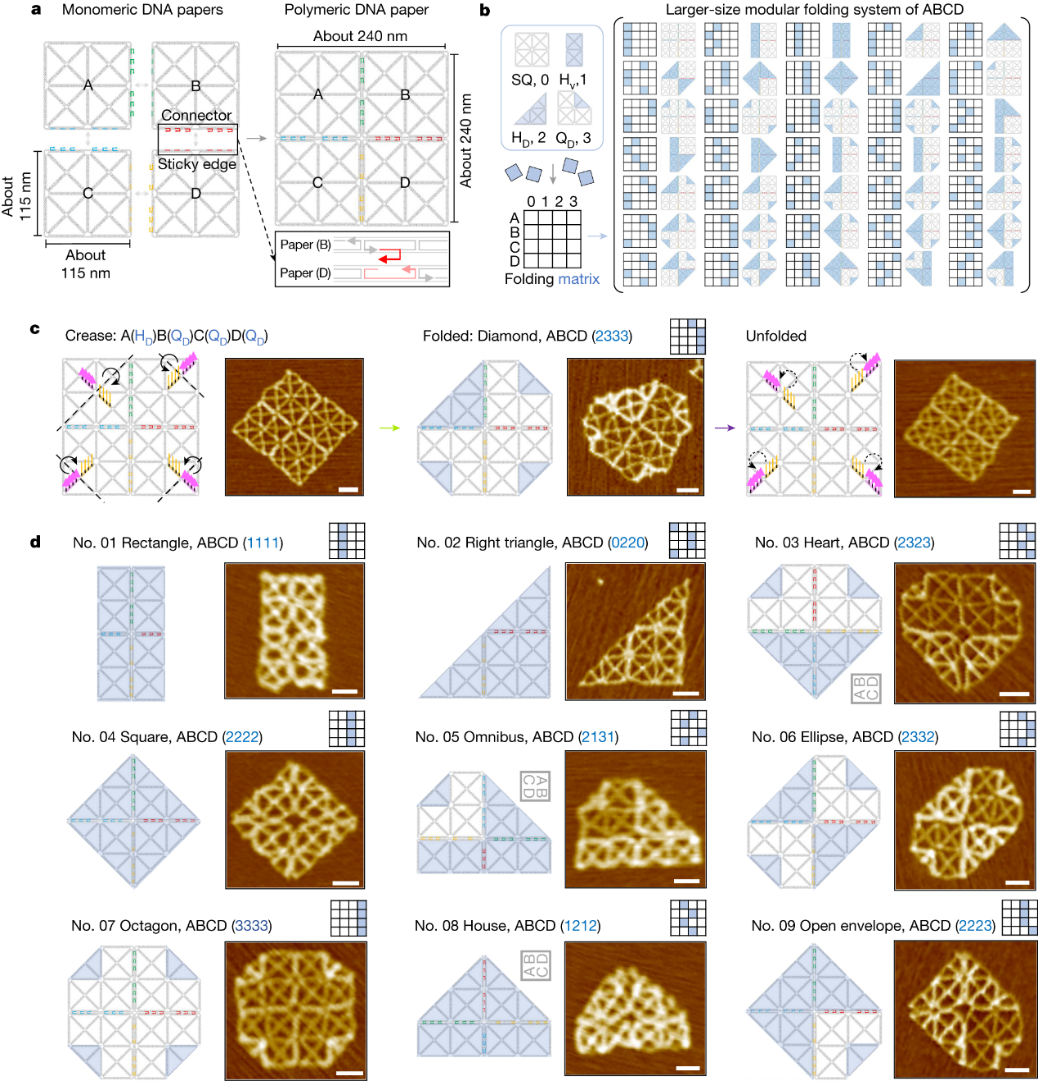
大尺寸可编程DNA线框纸折叠。图片来源:Nature
尽管折纸因娱乐性和美学而广为人知,但它也越来越多地被应用于工程技术中。纳米级折纸更有望用于纳米机器人、超灵敏分子传感、超材料等领域。“这项工作的设计独具匠心,核心想法简单且美丽,像孩子折纸一样折叠DNA结构”,瑞士洛桑联邦理工学院Maartje Bastings说,“作者们还结合强度和分层组织的设计原理,对DNA折纸的构象变化、动力学、刚性和柔性之间的平衡提出了独到的见解”。“折纸艺术启发了DNA折纸的发展,通过可编程、可重构的纳米操作,作者设计了DNA线框结构,进一步的用途将令人期待”,Nature 杂志的高级编辑Magdalena Helmer如此评价 [4]。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Harnessing a paper-folding mechanism for reconfigurable DNA origami
Myoungseok Kim, Chanseok Lee, Kyounghwa Jeon, Jae Young Lee, Young-Joo Kim, Jae Gyung Lee, Hyunsu Kim, Maenghyo Cho & Do-Nyun Kim
Nature, 2023, 619, 78-86. DOI: 10.1038/s41586-023-06181-7
参考文献:
[1] P. Rothemund, Folding DNA to create nanoscale shapes and patterns. Nature 2006, 440, 297-302. DOI: 10.1038/nature04586
[2] 院士Talk•樊春海:我有一根长长长长长长的DNA,和一些短的DNA,你想要什么图案?我给你织!
https://www.bilibili.com/video/av90354202/
[3]新民周刊:樊春海:70后院士横跨三个领域
https://m.xinminweekly.com.cn/content/16014.html
[4] Nature News: Nanoscale origami with DNA-wireframe paper
https://www.nature.com/articles/d41586-023-02022-9
(本文由小希供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!































 京公网安备 11010802027423号
京公网安备 11010802027423号