释放Cas12a的全部潜力:底物和反应条件对反式切割活性的影响
注:文末有研究团队简介及本文科研思路分析
成簇规则间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats, CRISPR)系统可以通过CRISPR derived RNA(crRNA)快速靶向特定基因序列并激活Cas核酸酶的切割活性。由于高度的可编程性,CRISPR已被应用于基因编辑、细胞内成像、转录调控等领域。其中,Cas12a是广泛研究的CRISPR Cas酶,其在识别和切割靶序列后显示出对单链DNA(ssDNA)独特的反式切割活性,成为了科学家们关注的焦点。近日,华中科技大学吴曈勃副教授(点击查看介绍)课题组基于已有对CRISPR/Cas12a性质的探究,巧妙通过改变切割底物和反应条件,将Cas12a活性提升约50倍。相关成果近期发表在国际化学权威杂志Analytical Chemistry 上。
通常,Cas12a与预扩增方法结合使用,而Cas12a本身表现出较低的反式切割活性,对于未经预扩增的核酸检测限仅达到nM水平。预扩增需要较长的反应时间和复杂的设计从而限制了Cas12a的应用。为了克服上述问题,华中科技大学吴曈勃副教授团队探究了底物长度和反应条件对Cas12a反式切割活性的影响。
他们将不同长度修饰荧光团(FAM,F)和猝灭剂(BHQ,Q)的ssDNA作为报告分子进行探究,发现对应的Cas12a体系荧光上升速率在5-30 nt的底物长度下呈抛物线变化,并在15 nt时达到最大值。相较于常用的5-nt ssDNA,Cas12a切割15-nt ssDNA的反应速率显著提升,原因可能在于15-nt ssDNA与Cas12a正电结构域到切割位点之间的距离接近,其更易进入Cas12a切割口袋并产生信号。文献报道的Cas12a正电结构域与切割位点距离约为6-12 nt。ssDNA报告分子中的磷硫酰化修饰片段无法被Cas12a切割,Cas12a切割普通7-nt ssDNA与含两个磷硫酰化修饰的7-nt ssDNA的速率相近,而切割普通5-nt ssDNA的速率明显下降。这表明,报告分子的长度是决定Cas12a切割速率的决定性因素,符合上述猜测。
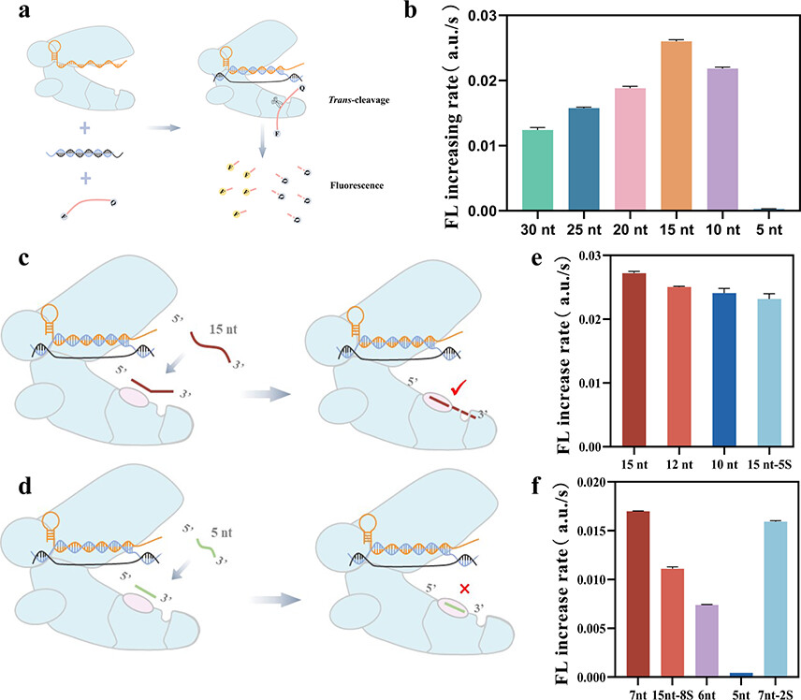
图1. Cas12a的反式切割活性。图片来源:Anal. Chem.
随后,作者在最佳底物长度的基础上继续探究反应条件对Cas12a活性的影响,发现Cas12a在NEBuffer 4中具有最好的反式切割活性,其对应的荧光上升速率比NEB与Cas12a一起提供的推荐缓冲液NEBuffer 2.1快近5倍。比较NEBuffer 2.1和NEBuffer 4成分,发现主要差异在于NEBuffer 4含有钾离子(K+)和二硫苏糖醇(DTT)。当在NEBuffer 2.1中添加额外的K+时,Cas12a的反应速率也会显著增加,而随着DTT浓度的增加,Cas12a的反应速率显著降低。因此K+可能促进了ssDNA报告分子进入Cas12a的活性中心。
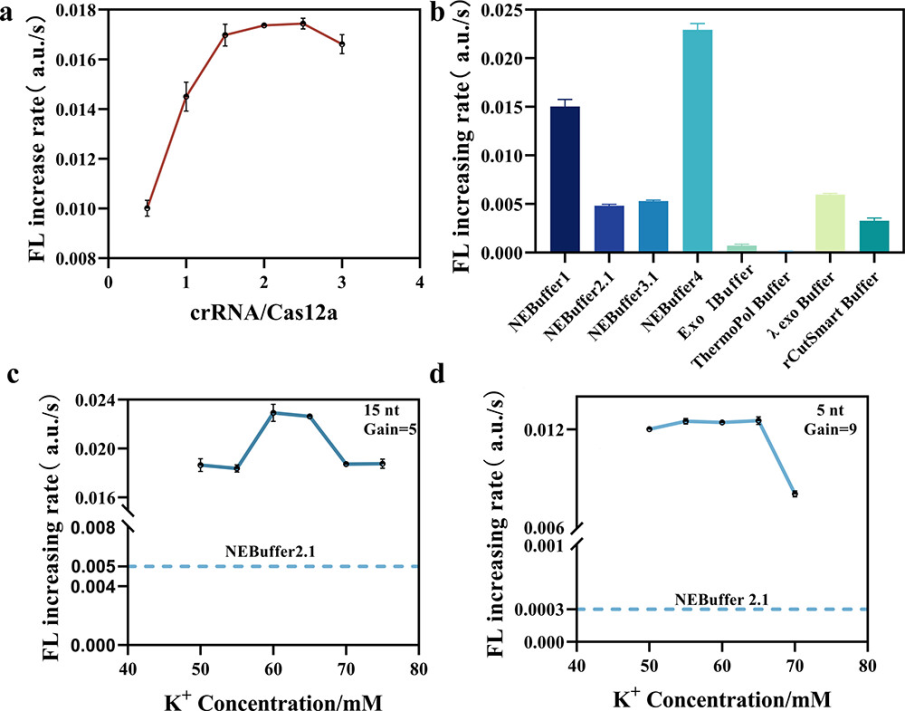
图2. Cas12a反应系统的优化。图片来源:Anal. Chem.
最后,论文测定了Cas12a系统针对不同双链DNA(dsDNA)或ssDNA底物的检测限(LOD)。在最优条件下(NEBuffer 4和15-nt ssDNA报告分子),dsDNA和ssDNA底物的LOD分别为0.12 pM和1.39 pM。而原始反应条件下(NEBuffer 2.1和5-nt ssDNA报告分子),dsDNA和ssDNA底物的LOD分别为0.17 nM和0.32 nM。因此,通过使用适当的底物和缓冲液可以将Cas12a的检测限降低1417倍和230倍。
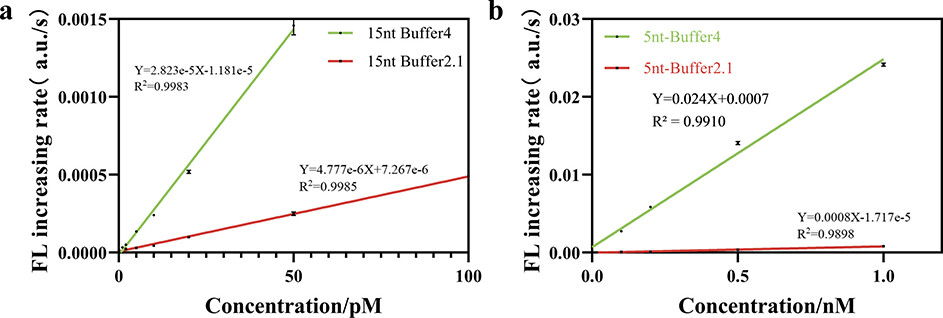
图3. Cas12a的检测限。图片来源:Anal. Chem.
该项工作通过简单改变报告分子长度和反应条件,可提高Cas12a反式切割活性近50倍,检测灵敏度提高2至3个数量级。与其他需要复杂设计的优化方法相比,工作方法操作简单,易于与其他预扩增方法衔接。
华中科技大学临床医学(八年制)专业本科生许杰和药学专业硕士研究生刘祝君为文章的共同第一作者,吴曈勃副教授为通讯作者。该研究工作得到了国家自然科学基金的支持。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Unlocking the Full Potential of Cas12a: Exploring the Effects of Substrate and Reaction Conditions on Trans-Cleavage Activity
Jie Xu,† Zhujun Liu,† Zhen Zhang, and Tongbo Wu*
Anal. Chem., 2023, DOI: 10.1021/acs.analchem.3c01307
吴曈勃副教授简介
华中科技大学同济医学院药学院副教授、博士生导师,湖北省高层次人才、武汉黄鹤英才。北京大学理学博士、理学学士、经济学(双学位)学士,加拿大阿尔伯塔大学博士后。主要研究兴趣为新型分子识别工具的开发及其在药物与精准医学分析中的应用。相关研究发表在Nat. Biomed. Eng.、Nucleic Acids Res.、Small、Chem. Sci.、Anal. Chem.等高水平期刊。
课题组主页:
https://www.x-mol.com/groups/Wu_Tongbo
科研思路分析
Q:这项研究最初是什么目的?或者说想法是怎么产生的
A:CRISPR/Cas12a一直是突变检测、核酸分析领域的研究热点,亦是我们课题组的研究兴趣之一。然而,Cas12a的应用往往受到其无扩增条件下较低活性的限制。因此,我们希望发展简便、通用的优化策略提高Cas12a的活性。
Q:研究过程中遇到哪些挑战?
A:我们在研究过程中遇到的最大的挑战是在发现报告底物长度对Cas12a体系灵敏度的显著影响后,如何科学、合理地猜测并证明产生该种变化的原理。尽管我们探究了5-30 nt ssDNA作为报告分子时Cas12a的活性差异,但长度变化与Cas12a活性改变之间的关系仍未可知。后来,我们猜测底物长度能够影响Cas12a切割速率的原因与Cas12a正电结构域到切割位点之间的距离有关。以上述假设为基础,我们通过磷硫酰化修饰的ssDNA与普通ssDNA作为报告分子进行比较,验证了上述猜想。
Q:该研究成果可能有哪些重要的应用?哪些领域的企业或研究机构可能从该成果中获得帮助?
A:该方法有望应用于CRISPR/Cas12a参与的各类检测体系中,简便、通用、有效的优化策略对其它CRISPR核酸酶灵敏度的提升也有一定参考价值。我们认为,在基因编辑、生物标志物检测等方向开展工作的企业或研究机构可能从中获得启发。
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!



















































 京公网安备 11010802027423号
京公网安备 11010802027423号