Nat. Chem.:时空可分辨全局性解析蛋白质-DNA相互作用新方法
蛋白质与DNA的相互作用在生物学过程中发挥关键作用,如转录因子(最重要的DNA结合蛋白种类之一)可特异性识别染色质开放区或可接近性的染色质DNA序列,精细地调控基因转录和表达。精确解析蛋白质-DNA相互作用能够揭示二者相互识别机制和动态变化,对于深入理解生理和病理条件下基因的调控机制至关重要。已有的研究方法,如高通量指数富集配体系统进化技术(HT-SELEX)、蛋白质结合微阵列(PBM)、DNA亲和沉淀-质谱分析(DNA-AP-MS)等,在表征高亲和力的DNA-蛋白质的识别和作用机制方面取得了很大的进展;然而对于生物体系中低丰度的蛋白质,以及动态、微弱的DNA-蛋白质(尤其是转录因子)复合物的分析仍极具挑战。
近日,中国科学院上海药物研究所陈小华(点击查看介绍)课题组与谭敏佳(点击查看介绍)课题组合作在Nature Chemistry 期刊发表研究论文,报道了一种解析蛋白质-DNA相互作用的新方法,结合质谱分析可发现与DNA探针相互作用的转录因子,能够时空可分辨解析低亲和力的转录因子-DNA相互作用;进而实现在不同条件下相关转录因子与特定序列DNA探针动态相互作用的解析,为全局性分析蛋白质-DNA的动态相互作用提供了一种新方法。
为了实现时空动态的蛋白质-DNA相互作用全景解析,研究团队在前期开发的光诱导PANAC光点击化学的基础上(Nat. Commun. 2020, 11, 5472; Chem 2019, 5, 2955-2968),发展了一种具有赖氨酸选择性的蛋白质-DNA交联方法(Light-Induced Lysine (K) Enabled Crosslinking, LIKE-XL),并结合团队在深度定量蛋白质组学分析方面的丰富经验和技术优势,实现了对蛋白质-DNA动态互作、包括弱相互作用的转录因子-DNA的时空动态性深度解析(图1)。
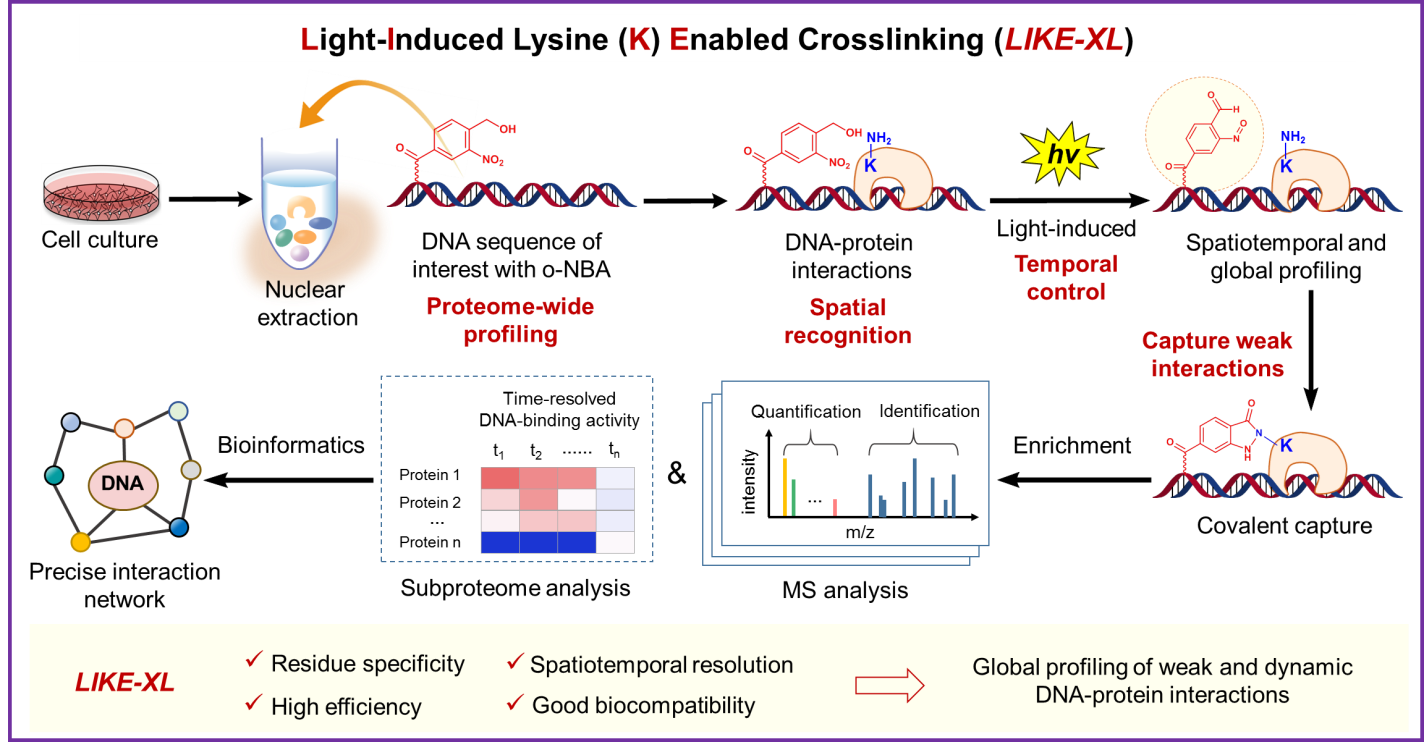
图1. LIKE-XL方法全局性解析蛋白质-DNA时空动态相互作用示意图
研究团队首先设计合成了含光交联基团的DNA探针,该探针能够在低浓度(微摩尔浓度)、短时间(5-10分钟)高效交联相互作用的蛋白质。结合基于质谱的定量蛋白质组学技术,LIKE-XL全局性解析方法在鉴定相互作用蛋白质数目上远超基于非共价作用的技术;比非特异性的交联方法显示更优的效率和更高的灵敏度,可获得更大的转录因子覆盖范围(图2)。
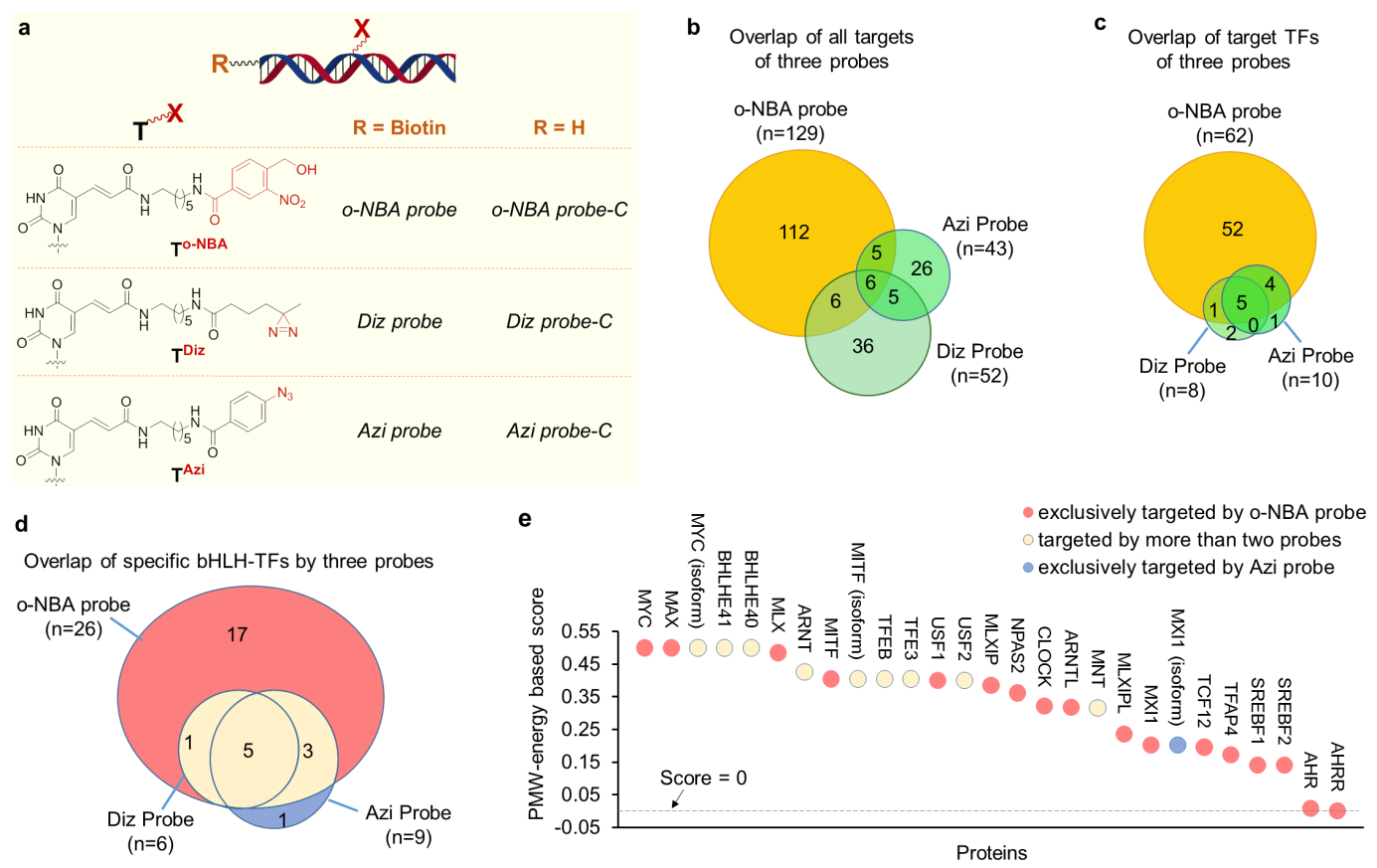
图2. LIKE-XL策略比非特异性的交联方法具有更优的效率和更高的灵敏度
LIKE-XL策略不仅成功地鉴定到了预期低亲和力的转录因子-DNA相互作用,还发现了目标DNA序列上新型转录因子及结合位点;通过后续测序表明新发现的转录因子能够富集相关的DNA序列。进一步,通过整合交联位点、结构生物学信息和分子对接,该研究发现了弱结合能力的转录因子与DNA结合的新作用模式(图3)。借助LIKE-XL技术,该研究全局性揭示了表观遗传药物(如去乙酰化酶抑制剂SAHA)时间分辨的下游转录因子互作网络动态全景。
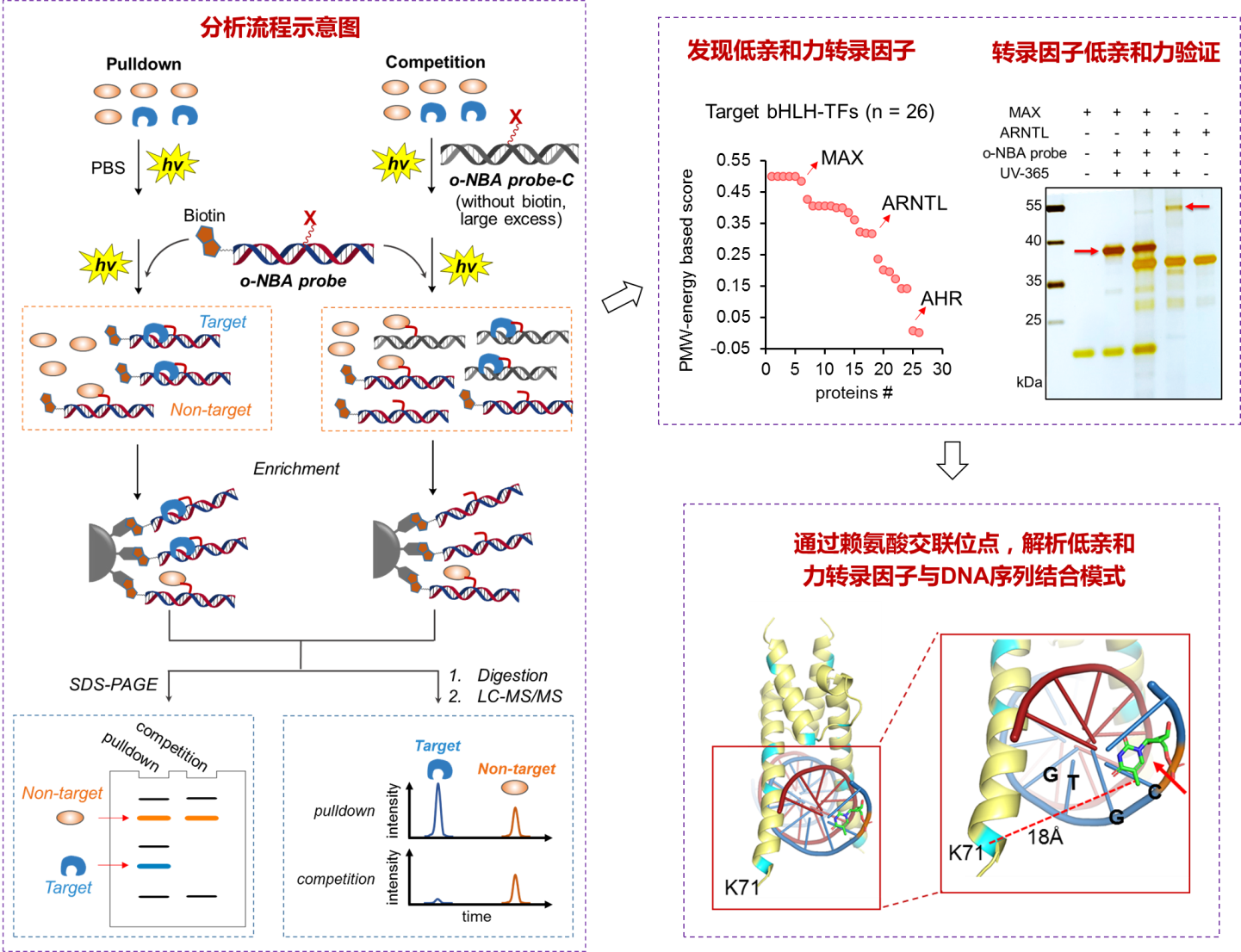
图3. LIKE-XL方法全局性解析与目标序列DNA相互作用的蛋白质,发现新低亲和力转录因子及新作用模式
总之,该项研究为全局性深度解析蛋白质-DNA的时空动态互作提供了一种新方法。LIKE-XL全局性解析策略,可用于以目标DNA序列为探针、时间可分辨解析相关转录因子的结合活性及动态互作变化的研究;在一定程度上可获得染色质开放区与转录因子识别的关键信息及探索相关生物学机制。以目标DNA序列为探针的互作蛋白质结合活性分析策略,有望用于解析目标DNA序列中不同的碱基修饰与蛋白质相互作用的研究,为DNA及表观遗传相关研究提供化学生物学新工具。
上海药物所陈小华研究员、谭敏佳研究员为本论文的通讯作者;郭安娣、闫克念、胡昊为本论文的共同第一作者。该工作得到国家自然科学基金委“生物大分子动态修饰与化学干预”重大研究计划、科技部重点研发计划等项目的支持。此项工作获得上海交大医学院张健教授,中科院上海药物所赵东昕研究员、陆晓杰研究员、许叶春研究员,江南大学徐勇将教授,复旦大学上海医学院刘贇教授的大力帮助和支持。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Spatiotemporal and global profiling of DNA–protein interactions enables discovery of low-affinity transcription factors
An-Di Guo, Ke-Nian Yan, Hao Hu, Linhui Zhai, Teng-fei Hu, Haixia Su, Yijia Chi, Jinyin Zha, Yechun Xu, Dongxin Zhao, Xiaojie Lu, Yong-Jiang Xu, Jian Zhang, Minjia Tan,* Xiao-Hua Chen *
Nat. Chem., 2023, DOI: 10.1038/s41557-023-01196-z
导师介绍
陈小华
https://www.x-mol.com/university/faculty/36593
谭敏佳
https://www.x-mol.com/university/faculty/36598
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!
-
Nat. Chem.:活细胞中“捕捉”DNA G-四链体相互作用蛋白质组 2021-06-29
-
新型探针捕捉DNA-蛋白质相互作用,解析DNA损伤应答 | 雷晓光课题组NSR 2023-02-07
-
活性共价化学标记-质谱分析蛋白质复合物局部微观结构 2017-03-29
-
Spatiotemporal and global profiling of DNA–protein interactions enables discovery of low-affinity transcription factorsAn-Di Guo, Ke-Nian Yan, Hao Hu, Linhui Zhai, Teng-Fei Hu, Haixia Su, Yijia Chi, Jinyin Zha, Yechun Xu, Dongxin Zhao, Xiaojie Lu, Yong-Jiang Xu, Jian Zhang, Minjia Tan, Xiao-Hua Chen
-
Thermal Proteome Profiling Reveals Insight to Antiproliferative and Pro-Apoptotic Effects of Lagunamide A in the Modulation of DNA Damage RepairYudong Hu, Dietrich Mostert, Christina Orgler, Oliver Andler, Hans Zischka, Uli Kazmaier, Angelika M. Vollmar, Simone Braig, Stephan A. Sieber, Stefan Zahler
-
A 35-bp Conserved Region Is Crucial for Insl3 Promoter Activity in Mouse MA-10 Leydig Cells.International Journal of Molecular Sciences (IF 4.9) Pub Date : 2022-12-01 ,DOI:10.3390/ijms232315060Xavier C Giner,Kenley Joule Pierre,Nicholas M Robert,Jacques J Tremblay
-
A brief review of protein-ligand interaction predictionComputational and Structural Biotechnology Journal (IF 4.4) Pub Date : 2022-06-03 ,DOI:10.1016/j.csbj.2022.06.004Lingling Zhao, Yan Zhu, Junjie Wang, Naifeng Wen, Chunyu Wang, Liang Cheng
-
Homodimeric and Heterodimeric Interactions among Vertebrate Basic Helix–Loop–Helix Transcription FactorsInternational Journal of Molecular Sciences (IF 4.9) Pub Date : 2021-11-28 ,DOI:10.3390/ijms222312855Ana Lilia Torres-Machorro
-
A Bit Stickier, a Bit Slower, a Lot Stiffer: Specific vs. Nonspecific Binding of Gal4 to DNAInternational Journal of Molecular Sciences (IF 4.9) Pub Date : 2021-04-07 ,DOI:10.3390/ijms22083813Thomas Carzaniga, Giuliano Zanchetta, Elisa Frezza, Luca Casiraghi, Luka Vanjur, Giovanni Nava, Giovanni Tagliabue, Giorgio Dieci, Marco Buscaglia, Tommaso Bellini


































 京公网安备 11010802027423号
京公网安备 11010802027423号