Coord. Chem. Rev.:基于报告基因系统对RNA剪接的检测、成像和应用
RNA剪接(RNA splicing)是指从pre-mRNA中去除内含子连接剩余外显子产生功能成熟mRNA的过程。RNA剪接是基因表达的关键步骤,剪接异常往往会造成基因突变,产生功能失调的蛋白,从而导致特定疾病的发生。传统的RNA剪接检测技术通常集中于对剪接后产物的鉴定与分析,无法对剪接过程中的调控因子、作用元件以及时空分布进行动态地细节描述,还会伴随对检测对象造成损伤的潜在风险,在细胞和活体中对RNA剪接异常的检测与定量仍是一个挑战。近日,西安电子科技大学王福教授与韩国高丽大学Jong Seung Kim教授等在国际化学权威期刊Coordination Chemistry Reviews(中科院一区TOP,IF 24.8),在线发表了题为 “RNA splicing based on reporter genes system: Detection, imaging and applications” 的综述文章。该论文总结了基于不同类型的报告基因系统对RNA剪接过程的监测、成像与生物医学应用(图1)。
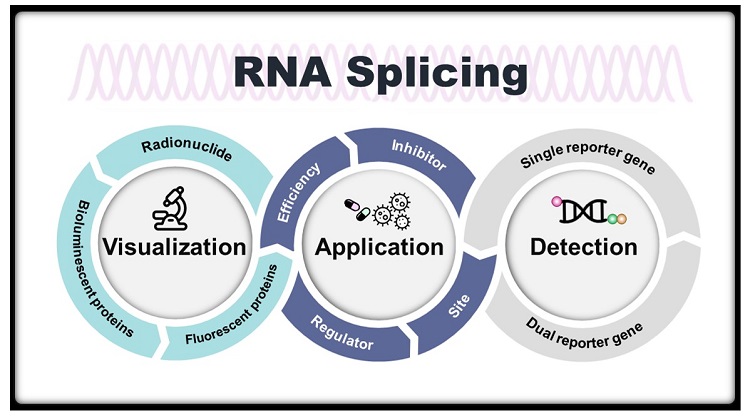
图1. 文章的内容概要
论文的第一部分,详细介绍了RNA剪接的机制、过程与类型。一般意义上的RNA剪接主要针对mRNA,多数mRNA前体内含子的两端边界具有同样的序列,遵从GU-AG法则。RNA剪接主要通过两步转酯反应来实现,并且是由高度动态的大型复合体剪接体所介导完成的(图2)。RNA可以选择性地越过某些外显子或某个剪切位点进行剪接,被称为可变剪接,主要包括外显子泄漏、外显子跳跃、外显子延伸和内含子保留等几种类型。除了上述主要的剪切方式,还有两种自剪接内含子——Ⅰ、Ⅱ类自剪接内含子,这些内含子本身具有催化活性,无须借助剪切体就可以进行内含子的自我剪接。此外还有Ⅲ类自剪接内含子与tRNA前体剪接内含子,都比较罕见。
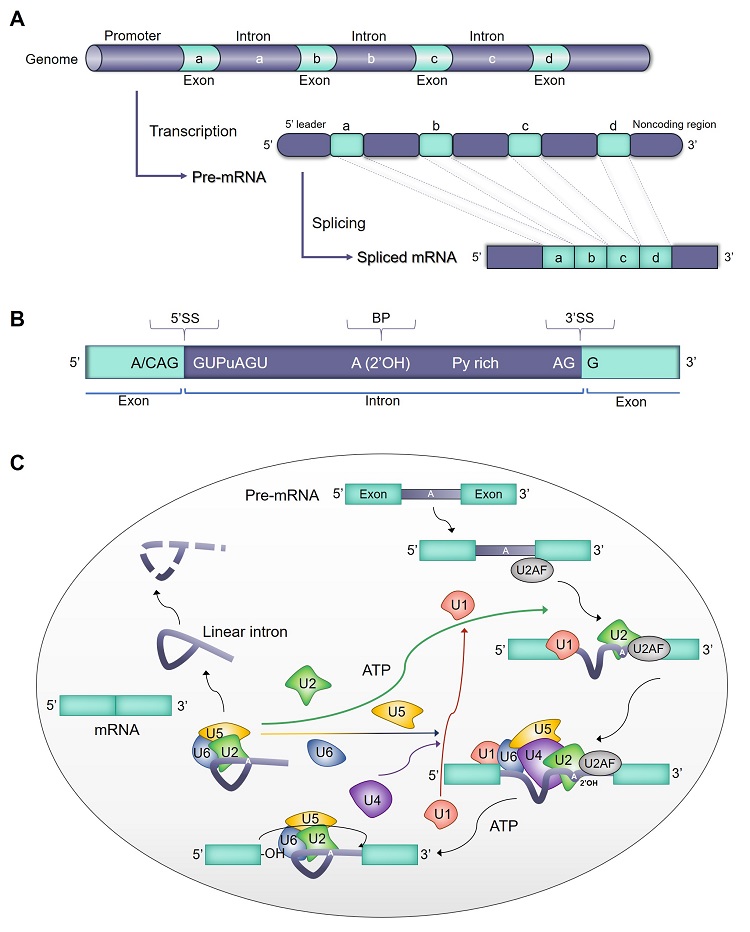
图2. RNA剪接的原理与过程
论文的第二部分,总结介绍了基因编码的报告系统用于RNA剪接的检测与成像,从所使用的报告基因的数目分为两大类--单报告基因系统与双报告基因系统。再根据所使用的报告基因的种类,分别介绍了基于荧光蛋白、荧光素酶、核酶、核素、HaloTag蛋白、MS2蛋白和剪接体介导的RNA反式剪接报告系统,对比了它们在检测、成像RNA剪接过程中的优缺点,探讨了它们潜在的应用价值与实际意义。论文的第三部分,归纳介绍了利用报告基因系统在RNA剪接方面的生物应用实例:可以对剪接过程所涉及的元件,如剪接调控因子进行筛选;对RNA剪接的效率实现定量评估;对特定的剪接位点进行筛选(图3);完成对剪接抑制剂或核酶变体的高通量筛选;以及实现对内含子在基因中具体位置的检测等。论文的最后,对未来基于报告基因系统用于RNA剪接的检测、成像和应用提出了展望,可以通过结合多个报告基因的优点,构建多基因融合的成像检测手段,来弥补目前报告基因在临床应用中的不足,通过结合现已批准的可安全用于人体的成熟报告基因,为临床RNA剪接相关疾病的诊断提供一种综合的多模态成像技术。
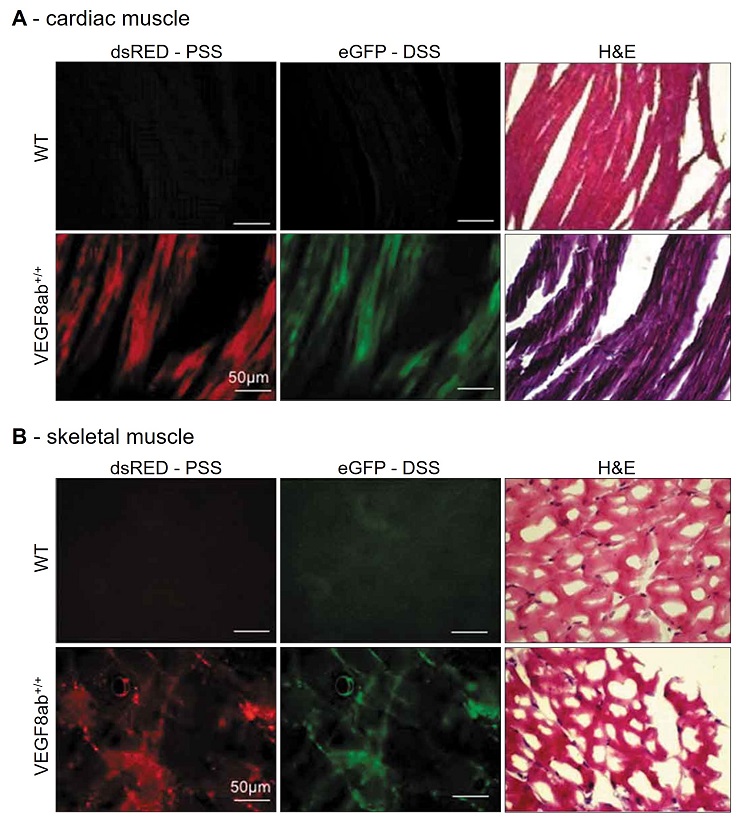
图3. 基于荧光蛋白的报告系统用于筛选RNA剪接位点
小结
报告基因系统通过其高效、灵敏的分子技术,为RNA剪接检测技术的发展提供了新的启发。通过报告系统对RNA剪接过程的无创动态成像,可以比较容易地确定靶基因的异常剪接与生命活动和疾病发生之间的密切关系,有望从科学实验走向临床应用。在该综述中,作者总结了近年来基于基因编码的报告系统在RNA剪接检测、成像与生物医学应用方面的进展,指出融合多种报告基因的多模态成像技术将是未来发展基于报告基因的RNA剪接检测技术的重要研究方向之一,将在人类疾病的诊断和靶向剪接小分子的识别中发挥至关重要的作用。
王福课题组的博士研究生施潇蕊、青年教师唐初副教授以及韩国高丽大学的Miae Won博士为该论文的共同第一作者,王福教授、印度科学与工业研究理事会Amit Sharma研究员以及韩国高丽大学Jong Seung Kim教授为该论文的共同通讯作者。西安电子科技大学为论文的第一作者单位。该文章得到了国家自然科学基金、陕西省杰出青年科学基金、韩国国家研究基金等项目的资助。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

RNA splicing based on reporter genes system: Detection, imaging and applications
Xiaorui Shi, Miae Won, Chu Tang, Qihang Ding, Amit Sharma, Fu Wang, Jong Seung Kim
Coord. Chem. Rev., 2022, DOI: 10.1016/j.ccr.2022.214929
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!


































 京公网安备 11010802027423号
京公网安备 11010802027423号