利用碱基编辑器实现基因组环形RNA的敲除 | Genome Biology论文
标题:Knockout of circRNAs by base editing back-splice sites of circularized exons
DOI:10.1186/s13059-021-02563-0
微信原文:点击阅读微信原文
近日,国际期刊Genome Biology在线发表了题为"Knockout of circRNAs by base editing back-splice sites of circularized exons"的研究论文,报道了利用碱基编辑器(Base editor, BE)实现环形RNA在基因组水平敲除的新策略。
环形RNA由前体RNA通过下游5'剪接位点与上游3'剪接位点反向剪接产生,其在人、小鼠、果蝇、植物等众多物种体内均有广泛表达。已有的研究表明,环形RNA的表达不仅具有高度的细胞/组织特异性,也与多种人类疾病(如癌症、系统性红斑狼疮和神经退行性疾病等)密切相关。但是,目前大部分环形RNA的详细功能及其作用机制不详,原因之一可能是缺乏高效和普适的环形RNA敲除方法。虽然已有报道利用CRISPR/Cas9技术删除成环序列或破坏外显子侧翼互补元件实现环形RNA在基因组水平的敲除方法,但是由于环形RNA的生成特性,基于上述CRISPR/Cas9技术的敲除方法仅在极个别的环形RNA上得以实现,因此亟需开发新的环形RNA敲除方法促进环形RNA的研究(Li et alMol Cell, 2018)。
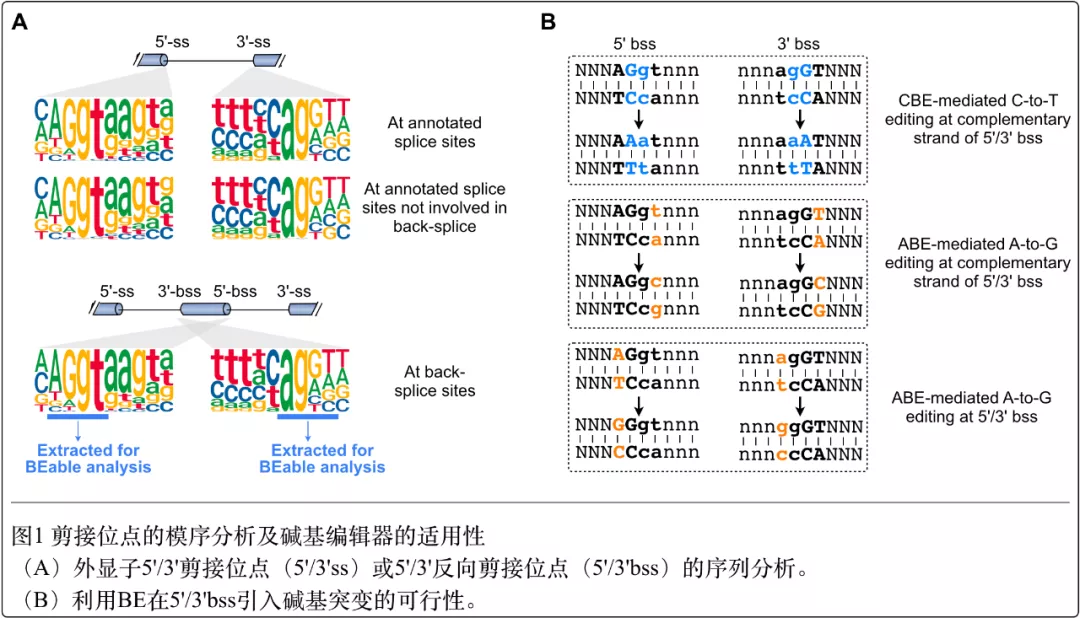
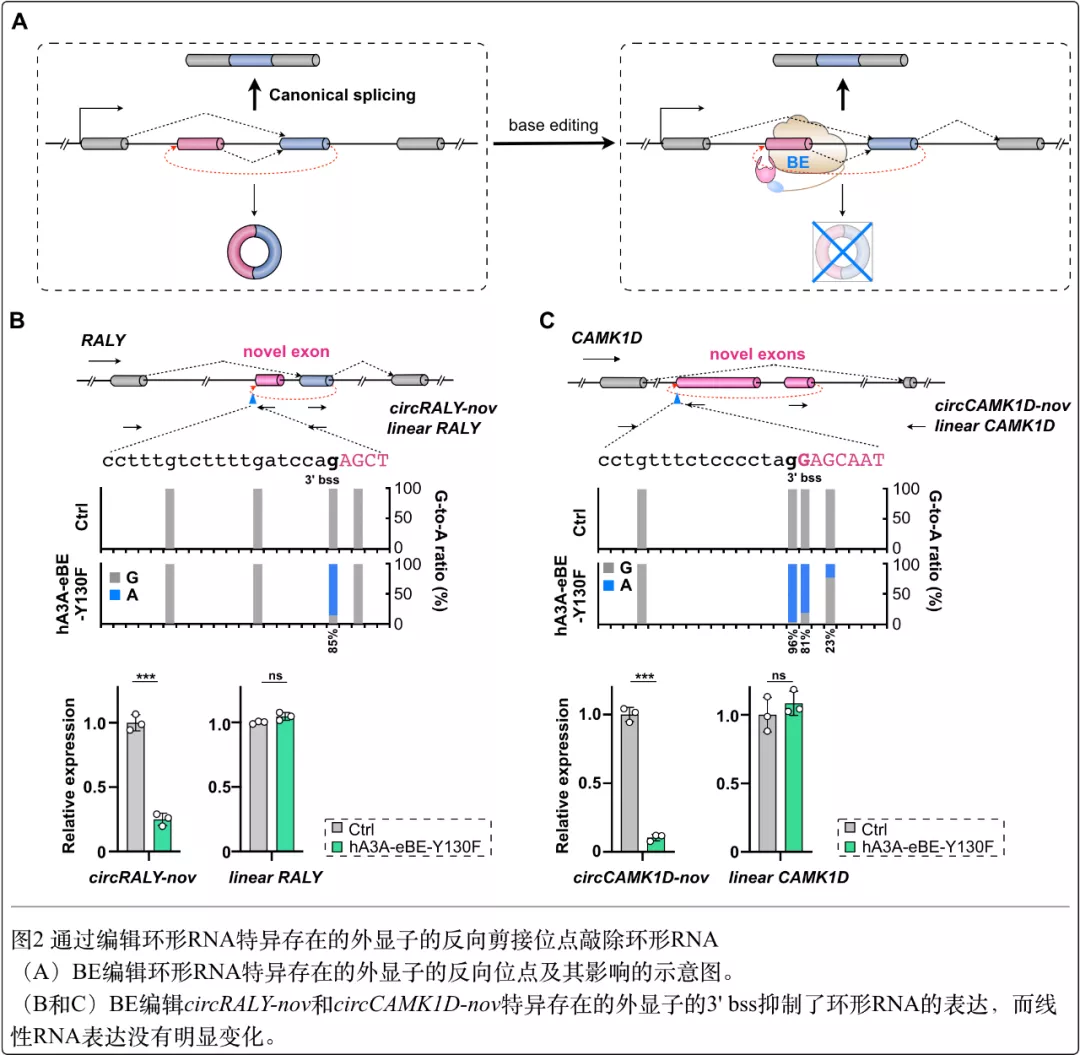
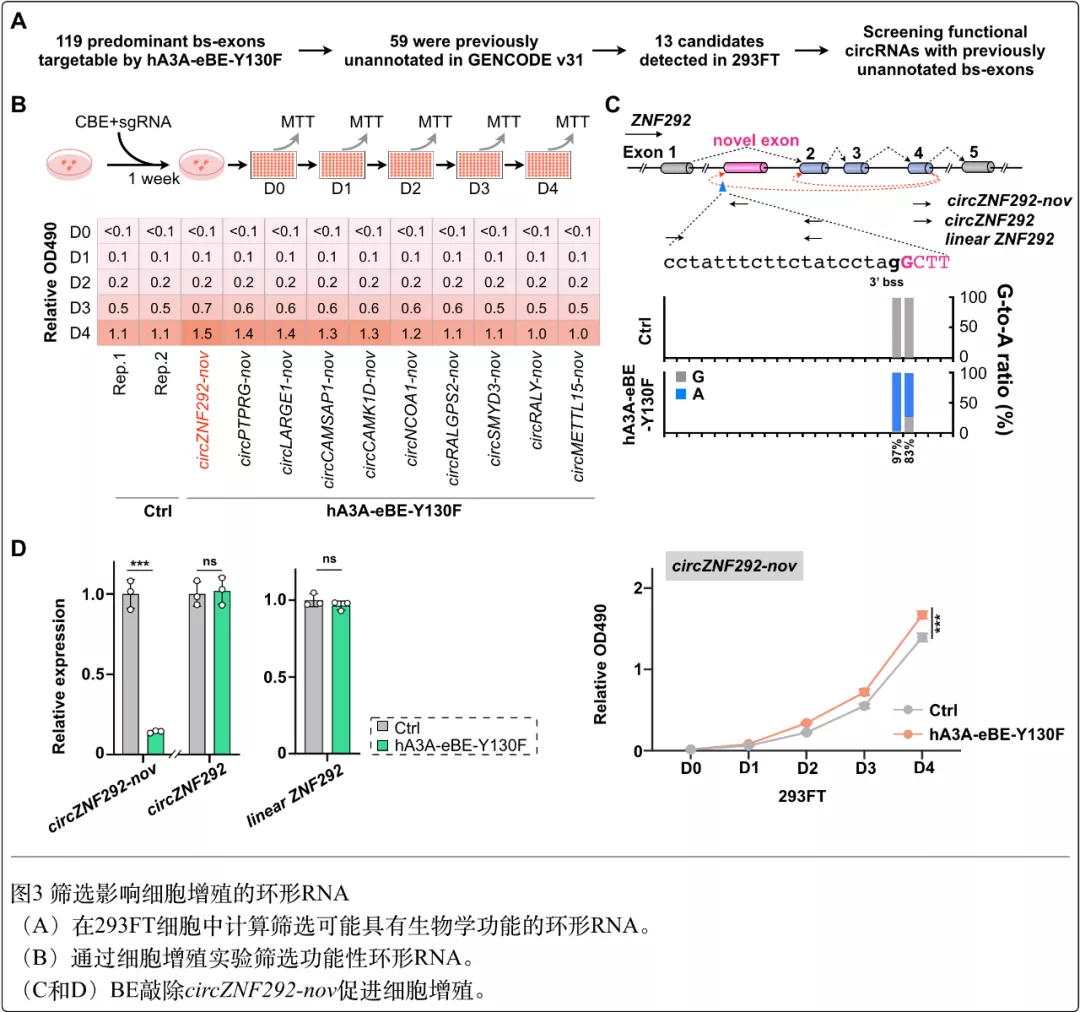
基于反向剪接位点具有与经典剪接位点(5'-AGgt---agGT-3')一致的固有模序序列这一特征,在这项最新的工作中,研究人员开发了利用碱基编辑器针对反向剪接位点开展单碱基突变的策略,实现了在基因组水平的环形RNA敲除新方法(图1)。一方面,研究人员利用CBE(实现反向互补链的C到T编辑)和ABE(编码链的A到G编辑)改变环形RNA及其同源线性RNA共用的剪接位点,发现其可以同时降低环形RNA和对应线性RNA的表达,首次在基因组水平证实了反向剪接和经典剪接之间的竞争。另一方面,研究人员针对只在环形RNA中存在的外显子反向剪接位点进行突变,实现了对环形RNA的特异性敲除。例如,针对已知环形RNA CDR1as/ciRS-7的反向剪接位点进行突变成功实现了CDR1as/ciRS-7的敲除,而对其相应线性RNA的表达影响微弱。通过计算分析,研究人员进一步筛选到一些只在环形RNA中特异存在的外显子,并利用BE对相应反向剪接位点进行突变,实现了针对环形RNA的特异敲除(图2)。重要的是,研究人员利用建立的新方法在293FT中筛选影响细胞增殖的功能性环形RNA,发现在ZNF292位点存在一个新的环形RNA分子circZNF292-nov,其含有一个仅在circZNF292-nov存在的外显子,而利用BE针对这个外显子反向剪接位点的突变实现了circZNF292-nov的敲除,其可以促进293FT的细胞增殖,提示circZNF292-nov具有抑制细胞增殖的潜能(图3)。这些研究结果证实了利用碱基编辑系统实现基因组水平环形RNA敲除新策略的可行性,也为开展环形RNA的功能探索提供了新的方法手段。最后,研究人员结合利用Cas9敲除(Zhang et al Cell Rep, 2016; Piwecka et alScience, 2017)或Cas13敲低(Li et al Nat Methods, 2021)环形RNA的方法,对如何利用BE开展环形RNA功能探索进行了讨论和展望,期望为后续的研究提供有益的参考和建议。
中国科学院上海营养与健康研究所(原中科院-马普学会计算生物学伙伴研究所)杨力研究员为该论文的通讯作者,中科院分子细胞卓越创新中心(原上海生化细胞所)和上海科技大学联合培养博士生郜祥和中国科学院上海营养与健康研究所博士后马旭凯为该论文的共同第一作者,中科院分子细胞卓越创新中心陈玲玲研究员和上海科技大学生命学院基因编辑中心陈佳教授为该工作的完成提供了大力帮助。该工作获得科技部、基金委、中科院、上海市科委和博士后基金等项目的资助。
参考文献:
Li S, Li X, Xue W, Zhang L, Yang LZ, Cao SM, Lei YN, Liu CX, Guo SK, Shan L, Wu M, Tao X, Zhang JL, Gao X, Zhang J, Wei J, Li J, Yang L, Chen LL. Screening for functional circular RNAs using the CRISPR-Cas13 system. Nat Methods. 2021;18(1):51-59.
Li X, Yang L, Chen LL. The Biogenesis, Functions, and Challenges of Circular RNAs. Mol Cell. 2018;71(3):428-442.
Piwecka M, Glažar P, Hernandez-Miranda LR, Memczak S, Wolf SA, Rybak-Wolf A, Filipchyk A, Klironomos F, Cerda Jara CA, Fenske P, Trimbuch T, Zywitza V, Plass M, Schreyer L, Ayoub S, Kocks C, Kühn R, Rosenmund C, Birchmeier C, Rajewsky N. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function. Science. 2017;357(6357):eaam8526.
Zhang Y, Xue W, Li X, Zhang J, Chen S, Zhang JL, Yang L, Chen LL. The Biogenesis of Nascent Circular RNAs. Cell Rep. 2016;15(3):611-624.
Genome Biology
doi:10.1186/s13059-021-02563-0
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!
































 京公网安备 11010802027423号
京公网安备 11010802027423号