UCR钟文婉课题组JACS:主客体识别联合机器学习实现非经典DNA结构分类与鉴别
DNA是储存遗传信息的生物大分子。它的碱基序列包含了指导蛋白合成的指令,同时它也可以形成不同的构象,比如经典的DNA双螺旋结构,及非经典的G-四链体(G4)、H-DNA、hairpin和i-motif结构等等。其中G4还可以分为不同的拓扑结构,即平行、反平行和混合型,并且G4中还可以存在凸起或者空位(图1)。H-DNA则根据其第三条链的走向,分为平行和反平行两种类型。DNA的折叠构象对DNA的稳定性和基因表达等都有调控作用。解析DNA的折叠构象有助于加深我们对其生物学功能的理解,并对开发靶向该结构的治疗手段具有指导意义。然而,DNA构象的多样性,某些结构的高度相似,及构象形成对溶液环境(比如缓冲溶液的pH、离子种类、浓度等)的要求,使得DNA构象分析充满了严峻的挑战。
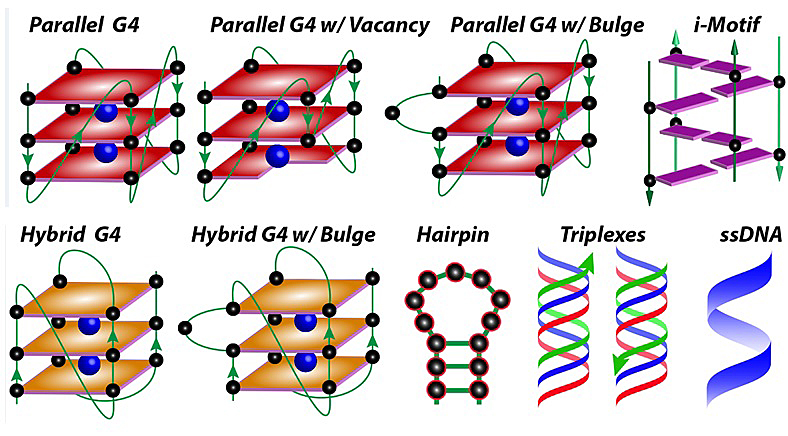
图1. 十种DNA非经典结构。图片来源:J. Am. Chem. Soc.
完整的DNA精细结构分析,依赖于强大、昂贵的仪器,像X-射线晶体衍射、多维核磁共振,并且耗时、消耗大量样品。圆二色谱可以用于简单的DNA二级结构分组,但是无法实现对于结构微小变化的分辨。荧光检测是潜在的简单方便又强大的方法,可以用于核酸结构的检测和分析。已经开发的有特异性识别G4的荧光分子,但是对于其他结构比如H-DNA和i-motif则少有能够特异性检测的荧光小分子,况且单一分子也很难区分结构亚型。
加州大学河滨分校(UCR)的钟文婉教授及Richard Hooley教授的合作团队首次证明了,由化学超分子组成的主客体阵列,可以通过荧光分析和模式识别的方法,对核酸结构进行快速准确的分类。他们2021年4月在Nature Chemistry 上发表的文章(Nat. Chem., 2021, 13, 488–495),阐述了运用主客体阵列对DNA G-四链体拓扑构象进行识别和分类。该阵列的优点是不需要针对DNA特定折叠构象的高度特异性染料分子,而是依赖于体系中多组分的差异性结合,利用DNA构象差异引起的荧光变化,对DNA结构进行辨别。
在这个工作的基础上,该合作团队继续发展多组分主客体识别阵列,对十种非经典DNA结构进行了分类和鉴别。他们最新的研究成果证明,将合成的超分子作为主体,荧光染料分子作为客体(图2),搭建的主客体识别体系可以高效分辨多种不同的DNA结构。DNA、主体分子、客体分子之间存在多种结合机制,这为针对DNA结构的指纹识别提供了基础。DNA的结构将决定DNA-主体-客体三者的结合关系,微小的改变将调整体系中的多重平衡关系,这种变化最终反映到荧光信号上。利用模式识别的统计方法对荧光信号的变化进行分析,从而达成对DNA不同构象的准确区分。这个方法甚至可以对高度相似的DNA结构的进行区分,比如G4和含空位或突起的G4、平行的H-DNA和反平行的H-DNA。

图2. 主客体分子结构。图片来源:J. Am. Chem. Soc.
利用模式识别的分析策略具有广阔的前景和应用价值,但是面对复杂的体系,它将产生大量的数据组,需寻求更强大的数据处理方法。机器学习是大数据分析的必要手段,已经被广泛应用于生物医药研究领域,包括生物信息、药物设计等方向,近来也被用于解决化学反应结果和机理分析等问题。机器学习的出色之处在于其能捕捉到隐藏在大量复杂嘈杂的数据背后的关联信息,通过训练模型,可实现对未知样的类别鉴定。
为了最大化地利用从感应分析体系中得到的大量数据,在这项最新的研究中,该团队使用了机器学习来处理阵列数据,实现分类十八条DNA序列结构的分类,并用其分类结果建立模型,成功预测了五条未知结构的DNA序列(图3)。
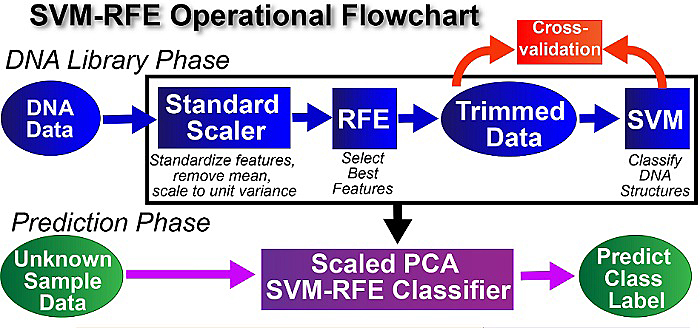
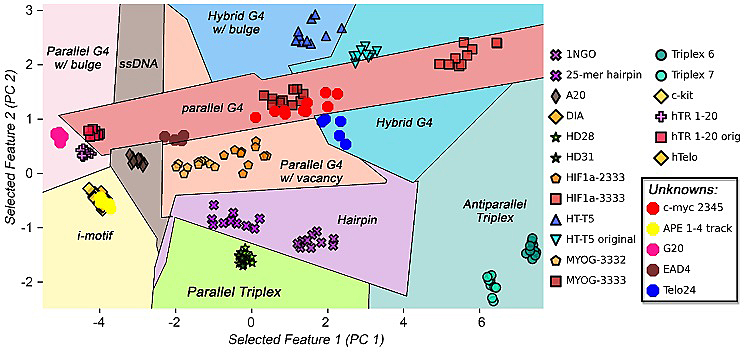
图3. 机器学习实现多种非经典DNA结构的分类及预测。图片来源:J. Am. Chem. Soc.
他们的实验结果说明,结合机器学习的手段,主客体阵列可以实现高特异性、高灵敏度、高准确率的DNA二维结构的分类和预测。设计对于特定折叠的高度特异性的荧光探针是一项高难度的工作,他们的方法可以巧妙地克服这一困难,解决问题的关键就是DNA的结构将决定DNA-主体-客体三者的结合关系,微小的改变将调整体系中的多重平衡关系,这种变化将反映到荧光信号上。作者表示,将把这种方法应用于更多核酸结构的直接且快速的分析。
这一成果近期发表在Journal of the American Chemical Society 上,文章的第一作者为加州河滨大学环境毒理系博士研究生陈筠怡。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Machine Learning Aids Classification and Discrimination of Noncanonical DNA Folding Motifs by an Arrayed Host:Guest Sensing System
Junyi Chen, Adam D. Gill, Briana L. HickeyBriana L. Hickey, Ziting Gao, Xinping Cui, Richard J. Hooley*, and Wenwan Zhong*
J. Am. Chem. Soc., 2021, 143, 12791–12799, DOI: 10.1021/jacs.1c06031
钟文婉教授简介
钟文婉,加州大学河滨分校 (UC Riverside) 化学系教授,环境毒理研究生项目主任。本科、硕士毕业于中国科学技术大学,2003年获爱荷华州立大学分析化学博士学位,其后在洛斯阿拉莫斯国家实验室完成博土后研究工作。担任Analytical Chemistry, Journal of the American Chemical Society, Chemical Reviews等重要学术期刊杂志的审稿人; 2019-2021担任美国国家科学基金会评审小组成员; 美国国家自然科学基金和香港研究委员会特别评审专家;主持了多个国际会议生化分析分会。曾获美国国家自然科学基金青年基金奖。主要研究领域涉及: (1) 发现有效的生物标记物和开发目标分子的快速、现场检测新技术和装置。(2) 研究纳米材料-生物分子界面用于生物功能化纳米材料的设计和应用。研究工作在Nature Chemistry; Journal of American Chemical Society; Angewandte Chemie International Edition; Chemical Sciences; Nucleic Acids Research; Analytical Chemistry等国际权威期刊上发表。
实验室主页:
https://faculty.ucr.edu/~wenwanz/index.html
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!
-
超分子指示剂竞争分析实现多色DNA荧光传感器 2019-02-10
-
高性能主-客体有机磷光材料及其在时间分辨生物成像中的应用 2021-04-17
-
三维微结构的数字微流芯片用于并行多种DNA分析 2017-06-30


































 京公网安备 11010802027423号
京公网安备 11010802027423号