DNA折纸结构力学探针助力基因分析
近日,中国科学院上海应用物理研究所物理生物学研究室与上海交通大学、南京邮电大学合作,基于DNA纳米技术发展了一系列DNA折纸结构并作为纳米力学成像探针,实现了原子力显微镜下对基因组DNA的直读检测和高分辨成像。相关结果发表在Nature Communications 上。
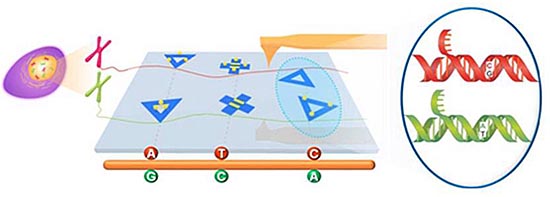
DNA折纸结构是利用DNA碱基互补配对原则,通过程序性设计将M13 DNA在上百条DNA短链的辅助下折叠成指定的几何形状。上海应用物理研究所张宏陆博士等在樊春海研究员的指导下与晁洁、师咏勇教授等合作,通过设计DNA折纸结构作为原子力显微镜的纳米力学成像探针,在单分子水平下实现了对DNA分子的特异性标记和单核苷酸变异性(SNP)的直读检测。相比于基于荧光成像的直读方法,这种新技术将分辨率提升了一个数量级,可达到远超光学衍射极限的10纳米分辨率。基于DNA纳米折纸结构设计的探针为原子力显微镜的图像获取提供了精确的标尺和丰富的选择,为遗传分析等生物学应用提供了新的工具。除此之外,他们还将该方法与之前发展的纳米PCR和单倍型分析技术(Nature Nanotechnology, 2011, 6, 639)结合,实现了单分子水平的遗传样本单倍型分析。这种单分子水平的单倍型分析通量高、可靠性好,有望用于易感基因的发现、疾病相关基因的鉴定和药物设计等方面。
该论文作者为:Honglu Zhang, Jie Chao, Dun Pan, Huajie Liu, Yu Qiang, Ke Liu, Chengjun Cui, Jianhua Chen, Qing Huang, Jun Hu, Lianhui Wang, Wei Huang, Yongyong Shi & Chunhai Fan
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

DNA origami-based shape IDs for single-molecule nanomechanical genotyping
Nat. Commun., 2017, 8, 14738, DOI: 10.1038/ncomms14738
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!






















































 京公网安备 11010802027423号
京公网安备 11010802027423号