“硅菌赛跑”,AI助筛超级抗生素
近年来,人工智能(artificial intelligence,AI)作为人类智慧的延伸,一直处于迅猛发展中。AI不但能在围棋、《星际争霸》等游戏中击败人类冠军,在工业和科研领域也有亮眼表现。比如,AI在皮肤癌诊断中准确度堪比专家(Nature, 2017, 542, 115,点击阅读详细),也能通过自主学习有机反应来设计分子合成路线(Nature, 2018, 555, 604,点击阅读详细),还可以与自动化合成“化学机器人”组合起来探索新反应(Nature, 2018, 559, 377,点击阅读详细)。
AI的强大能力也引起了药物研发科学家的注意,是否能用AI来帮助加速新药研发呢?比如,帮助新型抗生素的研发以解决日益严重的耐药菌问题。近日,美国麻省理工学院James J. Collins教授和Regina Barzilay教授等研究者训练一种深度神经网络(deep neural network)基于结构预测分子的抗菌活性。结合实验分析,他们发现了多种与已知抗生素在结构上相差较大的抗菌化合物,其中一种名为halicin的化合物对多种病原体都有优秀的杀菌活性,包括结核分枝杆菌和耐碳青霉烯肠杆菌。相关论文发表于Cell 杂志。

AI帮助筛选新型抗生素的流程示意图。图片来源:Cell
在这项研究中,他们首先从头(de novo)开始获取一个成本低廉、化学多样性高且不需要复杂实验室资源的训练数据集(training dataset)。他们选择美国食品药品监督管理局(FDA)核准药品库,再加入800个分离自动植物和微生物的天然产物分子,去重复后得到2335个分子。以对大肠杆菌E. coli BW25113的生长抑制达到80%作为筛选命中标准,初筛得到120个分子。接下来,他们对初级训练集的2335个分子按照初筛抑菌活性进行二值化(命中或未命中),利用这些数据训练出一个根据化合物的结构预测其是否抑制E. coli生长的二值化分类模型。为了达到这一目的,他们采用了一种定向信息传递的深度神经网络模型。该模型经过上述含2335个分子的训练数据集训练和优化后,应用到包括6111个分子的Drug Repurposing Hub数据库中以鉴别潜在的抗菌化合物。他们根据模型预测对化合物进行打分,并与其它模型进行了比较。他们从Drug Repurposing Hub中筛选出99个最有可能具有抗菌活性的分子,并对这些分子进行E. coli生长抑制实验,从中观察到51个分子对E.coli有生长抑制作用。同时,他们挑选得分最低的63个分子重复上述实验,只有2个分子有抑制作用。这反映了模型预测得分与生长抑制作用正相关。
在有活性的51个分子中,他们优先考虑处于临床前(Pre-Clinical)或1/2/3期临床研究且与训练集分子结构相似性较低、预测毒性低的化合物。符合所有这些标准的化合物是halicin,这是一种与训练集分子结构相似度低、尚处于临床前研究阶段、用于治疗糖尿病的硝基噻唑化合物(下图H)。
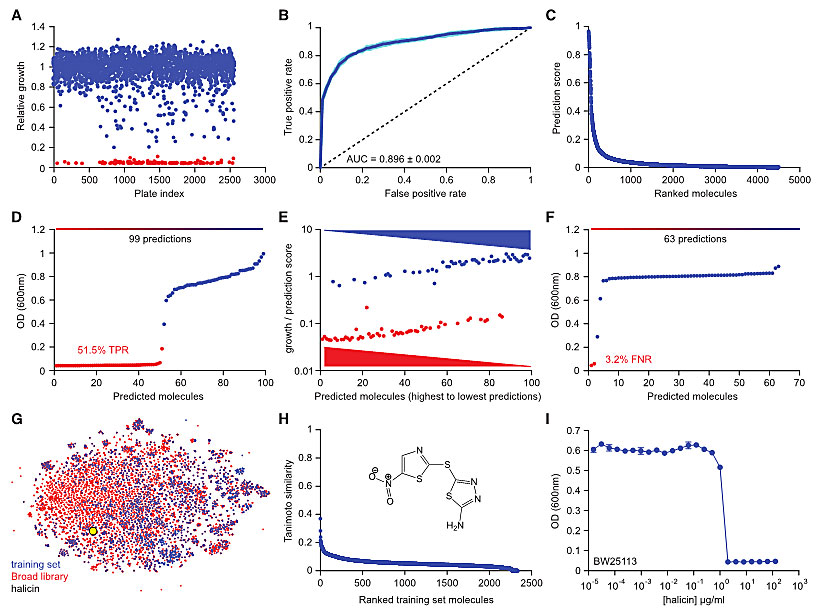
模型训练及halicin的发现。图片来源:Cell
为分析halicin的抗菌机制,他们进行了halicin的时间和浓度依赖性的杀灭实验,以确定化合物是通过杀菌(bactericidal)还是抑菌(bacieriostatic)机制来抑制细菌生长。他们观察到在营养丰富的生长条件下,halicin的存在对细菌细胞存在杀灭作用。随后,他们考虑了在代谢抑制、抗生素耐受条件下,halicin是否会诱导E. coli的死亡。结果表明,halicin对处于代谢抑制条件下的E. coli依然有杀灭作用,这与氨苄西林只能对付代谢活跃细菌形成鲜明对比。Halicin对一系列含抗生素耐药性基因(耐多粘菌素、氯霉素、β-内酰胺、氨基糖苷类、氟喹诺酮类、硝基呋喃类抗生素)的E. coli依然有抗菌活性。
为了更全面评估halicin抑制耐药菌的能力,他们检测了halicin对结核分支杆菌(Mycobacterium tuberculosis)、36株耐碳青霉烯类抗生素的肠杆菌(Enterobacteriaceae)临床分离株、鲍曼不动杆菌(Acinetobacter baumannii)和铜绿假单胞菌(Pseudomonas aeruginosa)临床分离株的生长抑制作用,这些病原体被世界卫生组织认定为急需新疗法的病原体。测试结果令人振奋,halicin对结核分支杆菌具有快速杀菌作用,对于肠杆菌、鲍曼不动杆菌和铜绿假单胞菌临床分离株具有较强的生长抑制活性。

Halicin的广谱杀菌作用。图片来源:Cell
Halicin对多种耐药菌的独特作用机制也引起了他们的兴趣。他们最初尝试通过抗halicin突变体进化分析来阐明其机制,然而无法分离到自发抑制的突变体。因此,他们应用RNA测序来了解E. coli的生理反应,用一系列浓度的halicin处理对数期细胞,并进行转录组测序。结果观察到所有浓度下和细菌细胞运动相关的基因明显下调,且在亚致死浓度下铁稳态所需基因上调。这促使他们推测halicin可能引起E. coli细胞膜质子动力的耗尽导致细胞的死亡,后续实验也支持了他们的推测。
鉴于halicin具有广谱杀菌活性,且不受质粒耐药元件或高频耐药突变的影响。他们接下来探索了该化合物在小鼠感染模型中的效果。为此,他们构建了由泛耐药型鲍曼不动杆菌感染伤口的小鼠模型,按照感染时间梯度用halicin对小鼠进行治疗。结果显示halicin对小鼠鲍曼不动杆菌感染模型有明显治疗效果。同时,他们把目光投向艰难梭菌(Clostridioides difficile)感染。这类病原菌会引起假膜性结肠炎,甲硝唑或万古霉素是临床一线治疗药物,在反复感染的情况下,现有抗生素显得无能为力,只有靠粪便微生物治疗来重新建立正常的结肠微生物群,但是这种治疗方式比抗生素治疗更具侵入性。在这项研究中,他们首先通过体外分析检测到halicin对C. difficile 630菌株有生长抑制作用,最小抑制浓度(MIC)为0.5 mg/mL。随后构建C.difficile感染的小鼠模型,以甲硝唑为阳性对照。结果发现,halicin对小鼠体内C. difficile 清除率强于甲硝唑,用halicin治疗后96小时,4只受试小鼠的C. difficile 被全部清除。
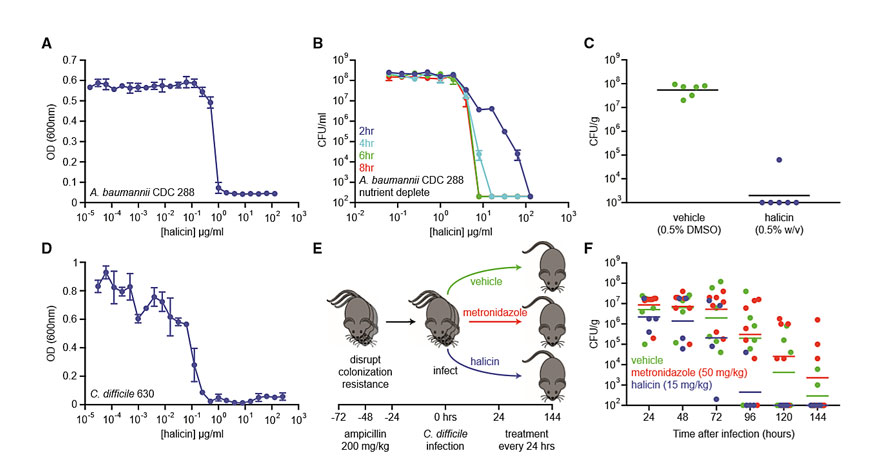
Halicin在小鼠感染模型中显示治疗效果。图片来源:Cell
在应用深度神经网络模型从Drug Repurposing Hub中预测出新抗生素候选药物halicin之后,他们又探索了两个化合物库——含9997个分子的WuXi抗结核库和含15亿分子的ZINC15数据库。他们首先利用Drug Repurposing Hub中收集到的经验数据对原模型进行重新训练,并将该模型应用于WuXi抗结核库,结果发现得分上限仅为0.37,这说明WuXi抗结核库中的化合物抗菌活性可能并不会太高。果然,他们将其中得分最高的200个化合物和得分最低的100个化合物进行整理和实验分析,结果所有300个化合物没有显示E. coli抑制活性。
在应用WuXi抗结核库中收集到的经验数据重新训练模型后,他们将模型投入到更大的ZINC15数据库中,并把关注点集中到其中约1.07亿个类抗生素分子。结果发现,有6820个分子得分大于0.7,3260个分子得分大于0.8,1070个分子得分大于0.9。他们根据模型预测得分对分子进行排序,并评估其与所有已知抗生素的Tanimoto相似性。由于他们对与现有抗生素结构不同的抗菌分子感兴趣,因此优先选择预测分数大于0.8且与任何已知抗生素的Tanimoto相似性小于0.4的化合物,这样筛选出23个符合标准的化合物进行实验。依次检测这些化合物对大肠杆菌、金黄色葡萄球菌、鲍曼不动杆菌、肺炎克雷伯菌和铜绿假单胞菌的生长抑制作用,结果从中发现了两个具有强广谱活性的化合物—ZINC000100032716和ZINC000225434673,二者与已知抗生素的相似性很低,并且其预测的人体潜在毒性较低。

从大规模化合物库中预测新抗生素。图片来源:Cell
虽然抗生素耐药性在全球范围内迅猛增加,但由于药物开发早期的高投入、高风险和长周期特点,具有临床应用价值的新抗生素生产力却显著下降。因此探索能够大幅度降低成本并提高抗生素发现成功率的新方法,对于重振抗生素候选新药研发生产流程至关重要。本文研究显示在解决这些障碍方面,AI具有很大潜力:(1)由于筛选只限于收集适当的训练数据,因此降低了先导化合物发现成本;(2)提高鉴定具有所需生物活性、结构新颖化合物的真实阳性率;(3)将寻找这些理想化合物所需的时间和劳力从数年或数月减少到数周。
希望在AI的助力下,这场与耐药菌的“硅菌赛跑”,人类能笑到最后。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

A Deep Learning Approach to Antibiotic Discovery
Jonathan M. Stokes, Kevin Yang, Kyle Swanson, Wengong Jin, Andres Cubillos-Ruiz, Nina M. Donghia, Craig R. MacNair, Shawn French, Lindsey A. Carfrae, Zohar Bloom-Ackerman, Victoria M. Tran, Anush Chiappino-Pepe, Ahmed H. Badran, Ian W. Andrews, Emma J. Chory, George M. Church, Eric D. Brown, Tommi S. Jaakkola, Regina Barzilay, James J. Collins
Cell, 2020, 180, 688–702, DOI: 10.1016/j.cell.2020.01.021
(本文由水村山郭供稿)
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!
































 京公网安备 11010802027423号
京公网安备 11010802027423号