“掌控进化的力量”,美英三科学家荣获2018年诺贝尔化学奖
10月3日,2018年诺贝尔化学奖揭晓,一半授予美国加州理工学院(Caltech)的Frances H. Arnold教授,获奖理由是“酶的定向进化”(“for the directed evolution of enzymes”),另一半由美国密苏里大学George P. Smith教授和英国MRC分子生物学实验室的Gregory P. Winter爵士分享,他们被表彰的贡献是“肽和抗体的噬菌体展示技术”(“for the phage display of peptides and antibodies”)。

从左至右:Frances H. Arnold教授、George P. Smith教授和Gregory P. Winter爵士。图片来源于网络
这三位科学家的研究方向差异不小,瑞典皇家科学院认为他们都“掌控了进化的力量”(“They harnessed the power of evolution”)。如今,公众已经普遍接受了进化论的观点,目前的生物多样性就是进化力量的充分体现。2018年诺贝尔化学奖的获得者们师法自然,基于相同的原理,采用巧妙的方法控制蛋白质的进化方向,这些成果极大增进了人类福祉。Frances H. Arnold教授的“定向进化”技术带来更高效更稳定的酶,作为生物催化剂用于更好地制造生物燃料、药物等。George P. Smith教授和Gregory P. Winter爵士的“噬菌体展示”技术带来的新抗体,可以用于治疗自身免疫疾病甚至某些转移性癌症。

酶的定向进化
Frances H. Arnold教授在1993年首次进行了酶的“定向进化”,从那时之后她不断努力改进这一技术,如今定向进化这一策略已经被广泛应用于生物以及生物交叉领域的各个分支,特别在酶工程这一方向上,有着无可替代的重要意义。
酶的定向进化与生物的进化十分类似,原理都是基因序列的改变以及后续的筛选。区别是酶的定向进化基因改变更迅速,后续的人工筛选更高效,这样可以让酶尽快达到预期的表现形式或者功能状态。这也是“定向进化”一词的由来——科学家控制着酶进化的方向。如下图所示,酶的定向进化大致分为四步:(1)在酶的基因中通过基因工程手段引入随机突变,(2)突变后的基因插入细菌,细菌将这些基因表达为蛋白质——也就是突变酶,(3)测试这些突变酶,一般是让它们催化目标化学反应,通过高通量方法快速、高效地筛选出那些表现最优的酶,(4)以筛选出的酶为模板,重复前三个步骤,如此循环,直到筛选出的酶达到要求。
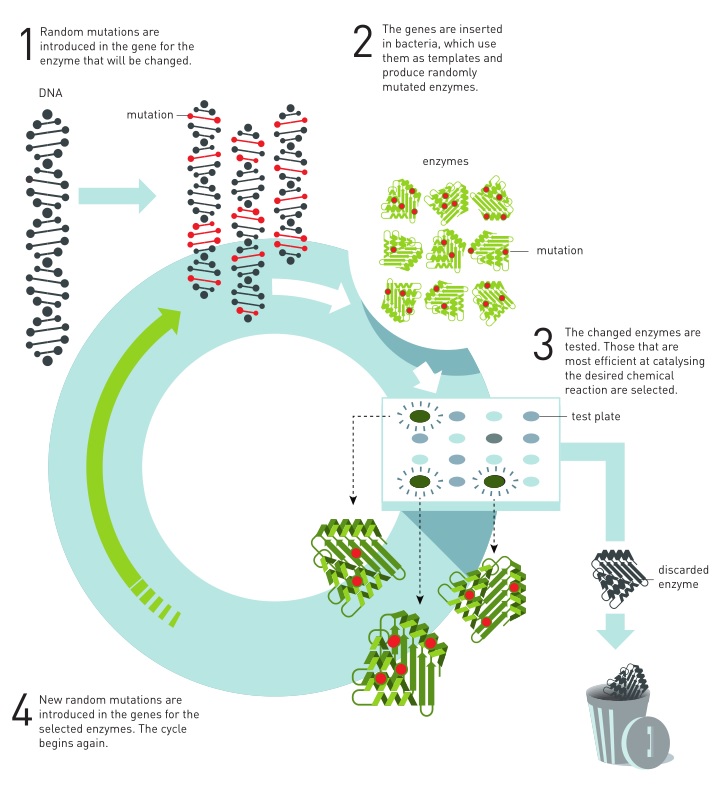
随着结构生物学研究的进步,人们对于酶的结构-活性关系的理解越来越深入,大量的酶结构被解析,关键的活性位点也一一被发现,这些成果使得科学家可以更有针对性地设计突变位点,而不是完全随机的进行突变,进一步提高了酶定向进化的效率和成功率。酶的定向进化会带来更稳定、更高效的生物催化剂,可以让药物及可再生燃料等物质的生产更加绿色环保并降低成本。除了提高酶的催化性能,定向进化还能赋予酶新能力,使它们可以催化那些原始酶在自然界中根本无法催化的反应。
X-MOL一直关注Frances H. Arnold教授团队,她们近些年的代表性工作包括:细胞色素c 用于碳硅键的构筑(Science, 2016, 354, 1048,点击阅读详细)和碳硼键的形成(Nature, 2017, 552, 7683,点击阅读详细),细胞色素P450用于烯烃的反马氏氧化(Science, 2017, 358, 215,点击阅读详细)、碳氢氨基化反应(Nat. Chem., 2017, 9, 627,点击阅读详细)以及合成传统有机合成实验室难以合成的高张力有机碳环体系双环丁烷和环丙烯(Science, 2018, 360, 71,点击阅读详细)。
噬菌体展示
噬菌体,可以看作一种能感染细菌的病毒。噬菌体和病毒一样,结构相当简单,由保护性的蛋白质外壳包裹着遗传物质(DNA或RNA)组成。当它们要扩增时,将遗传物质注入细菌并“劫持”它们的新陈代谢系统,让细菌生产噬菌体的遗传物质和蛋白质,并组装形成新的噬菌体。
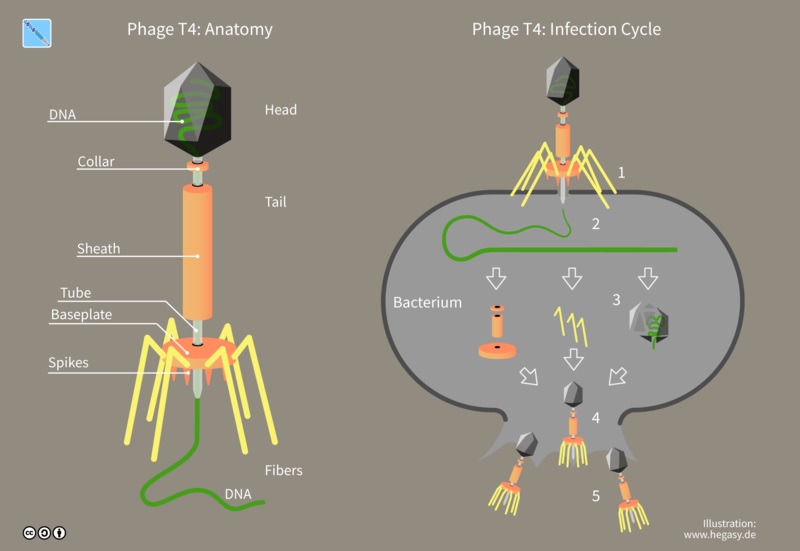
T4噬菌体及其感染细菌的过程。图片来源:Wikipedia
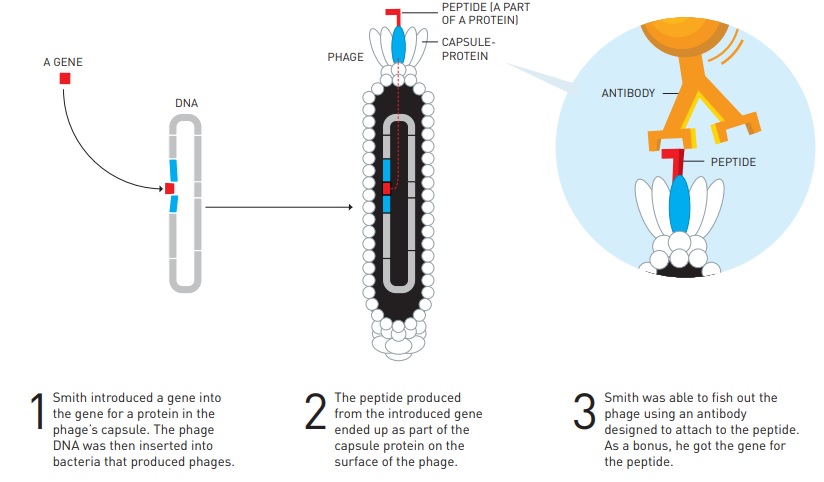
1985年,George P. Smith开发了一种称为“噬菌体展示”的技术,创造性地将噬菌体用于产生新蛋白质。噬菌体展示技术的基本原理如下图所示:(1)将编码蛋白质或多肽的外源基因插入噬菌体外壳蛋白基因,(2)噬菌体感染细菌进行扩增,插入的蛋白质或多肽与外壳蛋白融合表达,融合蛋白展示在噬菌体的表面,而外源多肽或蛋白则保持相对独立的空间结构和生物活性,可以与靶分子(例如抗体、受体、核酸、某些碳水化合物等)结合和识别,(3)分别导入了多种外源基因的一群噬菌体构成一个可展示外源蛋白质或多肽的噬菌体展示库,科学家可以用特定靶分子去筛选该噬菌体展示库,找到能选择性地同与其相互作用的某个噬菌体,也就找到了能与之结合的蛋白质和多肽及其编码基因。
Gregory P. Winter爵士创造性地使用噬菌体展示技术进行抗体的定向进化,原理也非常类似生物进化——基因序列的改变以及后续的筛选。抗体本质上也是一种蛋白质,定向进化过程包括数个“抗体基因随机突变——基于噬菌体展示技术筛选”的循环(具体如下图所示),直到获得想要的抗体。
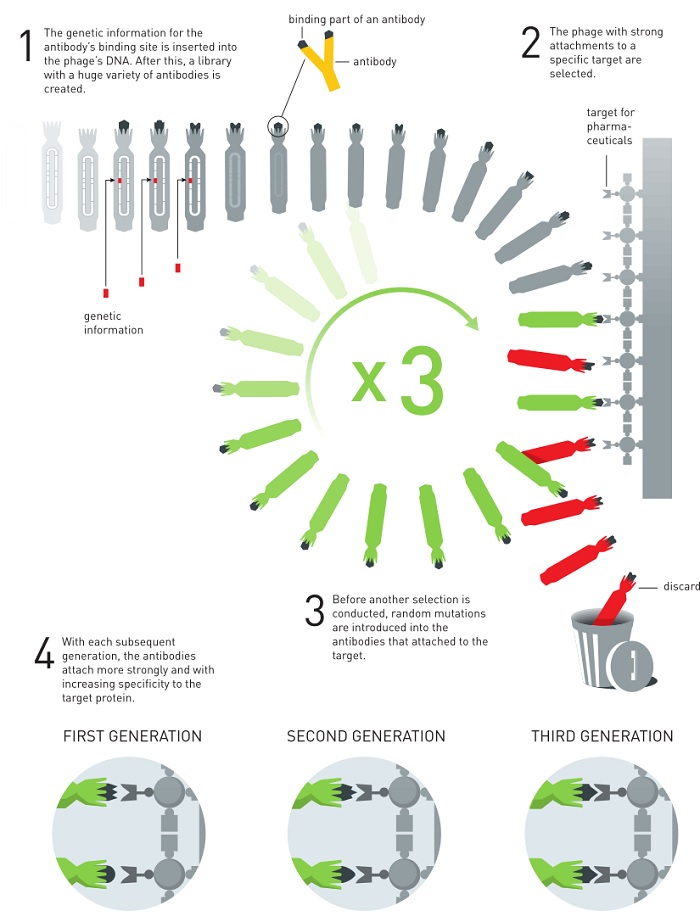
如此获得的抗体可以用做药物对抗一些普通化学药物难以对付的疾病。第一种基于这种方法得到的抗体药物是2002年获批上市的阿达木单抗(adalimumab),用于治疗类风湿性关节炎、银屑病和炎症性肠病。
噬菌体展示技术经过近20年的发展和完善,在生物医学领域还有其他十分广泛的应用,包括抗原表位分析、生物分子相互识别、疫苗研究、药物开发、病原检测、基因治疗、细胞信号通路研究等等。
关键论文
Chen K, Arnold FH. Tuning the activity of an enzyme for unusual environments: sequential random mutagenesis of subtilisin E for catalysis in dimethylformamide. PNAS, 1993, 90, 5618-5622.
Arnold FH. Engineering proteins for unusual environments. FASEB J. 1993, 7, 744-749.
Smith GP. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science, 1985, 228:1315-1317.
Parmley SF, Smith GP. Antibody-selectable filamentous fd phage vectors: affinity purification of target genes. Gene, 1988, 73, 305-318.
McCafferty J, Griffiths AD, Winter G, Chiswell DJ. Phage antibodies: filamentous phage displaying antibody variable domains. Nature, 1990, 348, 552-554.
注:以上内容编译自诺贝尔奖官方网站,图片等内容版权归属于Nobelprize.org
往年诺贝尔化学奖回顾:
2016诺贝尔化学奖:分子机器终折桂,CRISPR等明年再来?
“冷冻电镜”胜出,三位生物物理学家分享2017年诺贝尔化学奖
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!



















































 京公网安备 11010802027423号
京公网安备 11010802027423号