基因魔剪CRISPR/Cas9新进展——新型利器xCas9 3.7利刃出鞘
本文获“BioArt”授权转载,版权归“BioArt”所有,任何转载需获得“BioArt”授权。
前不久,哈佛与MIT的Broad研究所David Liu课题组在Nature发表的长文报道了一种升级版的Cas9新贵——xCas9,具有广泛的PAM兼容性和非常高的DNA特异性,此外还兼具剪切高效性等特点,堪称基因编辑领域又一款革命性工具。然而围绕该工具开发的一些背景以及其它相关工作,知道的人并不多。为了让读者更好的了解该工作的重要意义,BioArt特别邀请到了温州医科大学谷峰教授撰文详解相关工作的来龙去脉并进行了深度点评,以飨读者!

CRISPR/Cas系统是从原核生物中发现的一种防御外源性遗传物质入侵的自身免疫机制,主要分为三种类型:Type I、Type II和Type III。而常用的CRISPR/Cas9系统是由II型改造而来,具有核酸酶活性,并能够通过向导RNA的作用靶向性结合生物基因组任何区域的目的基因,进而实现对目标基因的编辑。该系统最早被开发并最成熟的是Streptococcus pyogenesCas9 (SpCas9),它具有较高的切割活性和保真性,是目前研究最深入的Cas酶之一。它识别的序列需要临近3’端有一段序列,即PAM(protospacer adjacent motif)。经典的PAM是NGG,而非经典PAM序列是NAG(N指 A/T/C/G)【1,2】。
在哺乳动物细胞中,SpCas9对靶位点在PAM为NGG时编辑效率是NAG的15倍;而中国水稻研究所王克剑教授团队对水稻基因组进行编辑时,则发现SpCas9在NAG位点的编辑效率约为NGG位点的76.44%,且保真性更高【3】。笔者利用人类细胞对应的报告系统对16个不同的PAM序列进行筛选后发现,SpCas9在部分位点对PAM为NGA的有效切割效率分别是NGG、NAG位点的0.5倍和2倍【4】。这项研究显示野生型SpCas9在人类细胞中可以利用其他非经典的PAM,但是效率并不高,也提示挖掘适用范围更广的SpCas9是有可能的。
古诗云:“长江后浪推前浪,浮事新人换旧人。”同样,在CRISPR的“舞台”上更是新秀迭出。
2018年1月29日Nature Biotechnology在线发表的文章A highly specific SpCas9 variant is identified by in vivo screening in yeast介绍了一种筛选SpCas9突变体的方法——酵母报告菌株筛选法。利用易错突变在SpCas9的REC3结构域处制造随机突变并构建突变体库,然后经酵母报告菌株(yACMO-off1–4)进行筛选并鉴别出较优突变体(图1)。研究者将筛选出的四个较优突变体进行组合突变,产生了最优突变体——evoCas9(evolved Cas9)。该突变体除了具有良好的靶向性和切割活性,它最突出的特点是超高的保真度,比之于野生型SpCas9提高了79倍【5】。鉴于此,evoCas9在基因编辑为基础的基因治疗方面将具有重要意义。
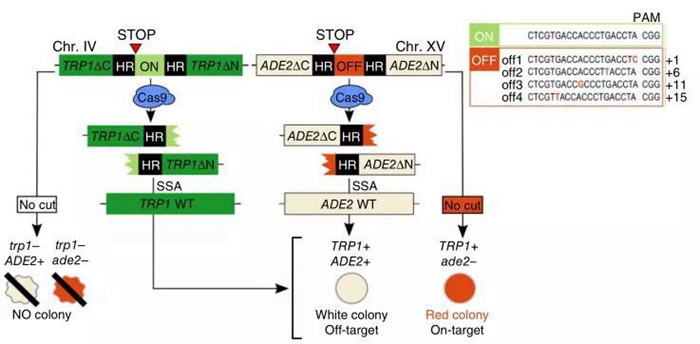
图1
无独有偶,2018年2月28日Nature在线发表了Evolved Cas9 variants with broad PAM compatibility and high DNA specificity。在该项研究中,为了更快速获得理想的SpCas9突变体,David Liu团队使用了独门秘籍——PACE(phage-assisted continuous evolution)(图2,图3)。其实早在2011年,Nature就刊登了David Liu 的另一篇文章A System for the continuous directed evolution of biomolecules,介绍了PACE定向进化技术【6】。该方法与以往在Cas酶编码基因序列中人为地制造突变,再利用原核/真核细胞进行功能筛选不同。PACE所使用的大肠杆菌含有诱导突变发生的质粒MP(mutagenesis)和激活后能表达PⅢ(噬菌体存活的必需基因)的质粒AP(accessory plasmid)。研究者们把需要突变的Cas酶蛋白编码基因序列放进噬菌体基因组,转染宿主大肠杆菌。PACE利用噬菌体繁殖周期短的特点,对Cas酶进行大批量、持续性的突变并定向筛选噬菌体。这项PACE技术通过把生物分子的实验室进化和噬菌体的生命周期结合在一起,一周内就能够实现成百上千次的传代变异,在诱变效率上比传统方法提高了100倍。
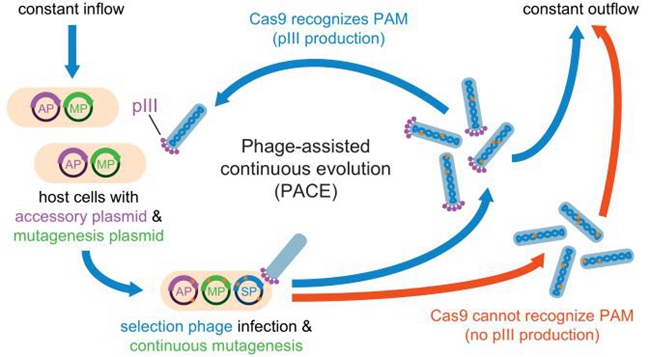
图2
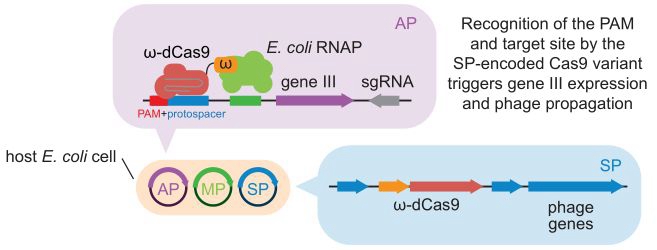
图3
在PACE技术的助攻下,突变体CRISPR/xCas9 3.7的出现,如雨后春雷,给基因编辑的应用发展带来了历史性的变革。比之于SpCas9,由SpCas9进化而来的xCas9 3.7有着更多的潜力。第一,xCas9 3.7具有更灵活的PAM选择性,能识别更大范围的PAM序列,包括NGG、NG、GAA和GAT;第二,它具有更优的靶向转录激活性和更强的切割基因的能力;第三,新构建单碱基编辑器xCas9(3.7)–BE3能够对致病性突变位点进行C•G→T•A和A•T→G•C的碱基替换,其效率分别高达73%和71%,明显优于SpCas9–BE3;第四,xCas9 3.7表现出极强的特异性,在PAM为NGG的EMX1基因靶位点处甚至没有脱靶。但令人困惑不解的是,xCas9 3.7的PAM序列识别灵活性、酶活性和保真性同时得到优化的现象突破了以往三者之间存在某种制衡的概念。目前这项工作仍然留下了不少需要回答的问题。另外,文献里报道的这些突变体(包括之前报道的基于结构为基础的突变体,如eCas9)平行比较实验也需要进行,如此可让大家了解这些工具,更好地选择最合适的工具。
实现高效的单碱基编辑一直是研究者们孜孜以求的目标,David Liu实验室在2017年报道过一种应用于哺乳动物细胞新型的腺嘌呤碱基编辑器ABE,借此编辑器实现A•T→G•C的转变【7】,当然其编辑效率远低于xCas9(3.7)–BE3。而中国科学院上海植物逆境生物学研究中心朱健康研究组在水稻中开发了一种新的腺嘌呤碱基编辑器ABEP2,其识别靶位点依赖于SaCas9(PAM序列NNGRRT),能够对水稻基因组的多个目标基因位点同时进行高效的碱基替换,甚至某些位点的编辑效率达61.3%,而且ABEP2精确性也很高【8】。总之,单碱基编辑器对精准编辑目的基因的研究意义非凡,就目前而言xCas9(3.7)–BE3无疑是 “利器”。
随着不同国家、不同地域的研究者们在CRISPR领域孜孜探索,人类基因组编辑技术的发展日新月异,新的方法和工具层出不穷。我们希望研究者们能够解析xCas9 3.7活性、保真性、PAM序列灵活性三者能够同时得到优化的机制,相信在此基础上,会有更快速、简便、高效、精准的基因编辑技术出现,被更广泛应用于疾病诊疗上、动植物育种等方面。但是,现有的技术水平距离人类基因编辑的需求仍有一定的距离,而xCas9 3.7(由SpCas9进化而来)的出现犹如利刃出鞘,将会极大地促进基因组编辑领域的进程。
这里需要提及的是,多种各具特色的Cas9中,在体内有优势的是SaCas9 (staphylococcus aureus Cas9)。2015年张峰实验室首先确定了SaCas9在哺乳动物细胞中的基因编辑作用,并对晶体结构进行了详细的分析。它的基因长度为3300bp,比SpCas9减少了约25%,这有助于SaCas9被载入腺相关病毒(AAV),增加了基因治疗应用的潜力;它识别的PAM是NNGRRT(N指A、T、C、G;R指A、G),在切割活性和靶向精准度上的表现均不输于SpCas9【9,10】。笔者实验室建立了双报告系统来评估基因组编辑工具的活性和PAM【11】,分别检测SpCas9、SaCas9和FnCpf1在不同位点上的基因组编辑活性,经过多重比较得出结论——SaCas9拥有比SpCas9和FnCpf1更高的活性【12】。因此,SaCas9凭借其分子量小和活性高的特点,在基因编辑应用中占据优势,更为简便高效。关于SaCas9的工程化改造可能对于基因组编辑为基础的临床治疗有重大意义。
在功能基因组学上,CRISPR家族新成员Cpf1因为能够同时完成多个基因的编辑,所以优势很明显【13】。国际上较为广泛采用的AsCpfl和LbCpfl对PAM序列有较为苛刻的要求(TTTN),极大程度上限制了Cpfl在实际应用中的靶位点的选择【14,15】。为了增加Cpf1靶序列的选择范围,笔者发现FnCpf1——其识别的PAM序列更加灵活(KYTV,K为G/T,Y为T/C,V为A/C/G),在人类细胞中具有较高的基因组编辑活性,这些为人类(含其他哺乳动物)基因组编辑提供了更多的工具【16】。如果能够进一步提高其保真性和活性,可能对于功能基因组学研究和临床疾病治疗非常有意义。另外,Cpf1家族对应的CrRNA较短,故而在工业化合成CrRNA方面更有优势。
综合上述,虽然xCas9 3.7的发现将让人类具有更加“锋利”和“通用性更强”的基因组编辑剪刀,但是它可能仍然无法做到基因组工具中“一统天下”,新的工具仍然需要研发和优化,从而实现“人定胜天”。
参考文献:
1. Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III.Nature, 2011, 471(7340): 602-607.
2. Hsu PD, Scott DA, Weinstein JA, Ran FA, Konermann S, Agarwala V, Li Y, Fine EJ, Wu X, Shalem O, Cradick TJ, Marraffini LA, Bao G, Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases.Nat Biotechnol, 2013, 31(9): 827-832.
3. Meng X, Hu X, Liu Q, Song X, Gao C, Li J, Wang K. Robust genome editing of CRISPR-Cas9 at NAG PAMs in rice.Sci China Life Sci, 2018, 61(1): 122-125.
4. Zhang Y, Ge X, Yang F, Zhang L, Zheng J, Tan X, Jin ZB, Qu J, Gu F. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells.Sci Rep, 2014, 4: 5405.
5. Casini A, Olivieri M, Petris G, Montagna C, Reginato G, Maule G, Lorenzin F, Prandi D, Romanel A, Demichelis F, Inga A, Cereseto A. A highly specific SpCas9 variant is identified by in vivo screening in yeast.Nat Biotechnol, 2018, 36(3): 265-271.
6. Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules.Nature, 2011, 472(7344): 499-503.
7. Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage.Nature, 2017, 551(7681): 464-471.
8. Hua K, Tao X, Yuan F, Wang D, Zhu JK. Precise A.T to G.C base editing in the rice genome.Mol Plant, 2018.
9. Nishimasu H, Cong L, Yan WX, Ran FA, Zetsche B, Li Y, Kurabayashi A, Ishitani R, Zhang F, Nureki O. Crystal Structure of Staphylococcus aureus Cas9.Cell, 2015, 162(5): 1113-1126.
10. Ran FA, Cong L, Yan WX, Scott DA, Gootenberg JS, Kriz AJ, Zetsche B, Shalem O, Wu X, Makarova KS, Koonin EV, Sharp PA, Zhang F. In vivo genome editing using Staphylococcus aureus Cas9.Nature, 2015, 520(7546): 186-191.
11. Yang F, Liu C, Chen D, Tu M, Xie H, Sun H, Ge X, Tang L, Li J, Zheng J, Song Z, Qu J, Gu F. CRISPR/Cas9-loxP-Mediated Gene Editing as a Novel Site-Specific Genetic Manipulation Tool.Mol Ther Nucleic Acids,2017, 7: 378-386.
12. Xie H, Tang L, He X, Liu X, Zhou C, Liu J, Ge X, Li J, Liu C, Zhao J, Qu J, Gu F, Song Z. SaCas9 Requires 5'-NNGRRT-3' PAM for Sufficient Cleavage and Possesses Higher Cleavage Activity than SpCas9 or FnCpf1 in Human Cells.Biotechnol J, 2017.
13. Zetsche B, Heidenreich M, Mohanraju P, Fedorova I, Kneppers J, DeGennaro EM, Winblad N, Choudhury SR, Abudayyeh OO, Gootenberg JS, Wu WY, Scott DA, Severinov K, van der Oost J, Zhang F. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array.Nat Biotechnol, 2017, 35(1): 31-34.
14. Zetsche B, Gootenberg JS, Abudayyeh OO, Slaymaker IM, Makarova KS, Essletzbichler P, Volz SE, Joung J, van der Oost J, Regev A, Koonin EV, Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system.Cell, 2015, 163(3): 759-771.
15. Zetsche B, Heidenreich M, Mohanraju P, Fedorova I, Kneppers J, DeGennaro EM, Winblad N, Choudhury SR, Abudayyeh OO, Gootenberg JS, Wu WY, Scott DA, Severinov K, van der Oost J, Zhang F. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array.Nature biotechnology,2017, 35(1): 31-34.
16. Tu M, Lin L, Cheng Y, He X, Sun H, Xie H, Fu J, Liu C, Li J, Chen D, Xi H, Xue D, Liu Q, Zhao J, Gao C, Song Z, Qu J, Gu F. A 'new lease of life': FnCpf1 possesses DNA cleavage activity for genome editing in human cells.Nucleic Acids Res, 2017, 45(19): 11295-11304.

如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!






















































 京公网安备 11010802027423号
京公网安备 11010802027423号