
- 从左至右,依次有原子编号,原子名,残基名,链名,残基号,原子坐标,occupancy,B-factor,元素名,segment name;
GUI操作
- 鼠标左键:rotate;中键:move;右键:zoom;
- file >> save session, 保存文件为pse或psw格式,可保存设置的各种效果,但无法再编辑;
- H>>hydrogens>>all/nonpolar #隐藏所有/非极性氢原子;
- 选定原子后,鼠标点击S可改变显示模式,H可隐藏显示模式,C可改变显示颜色
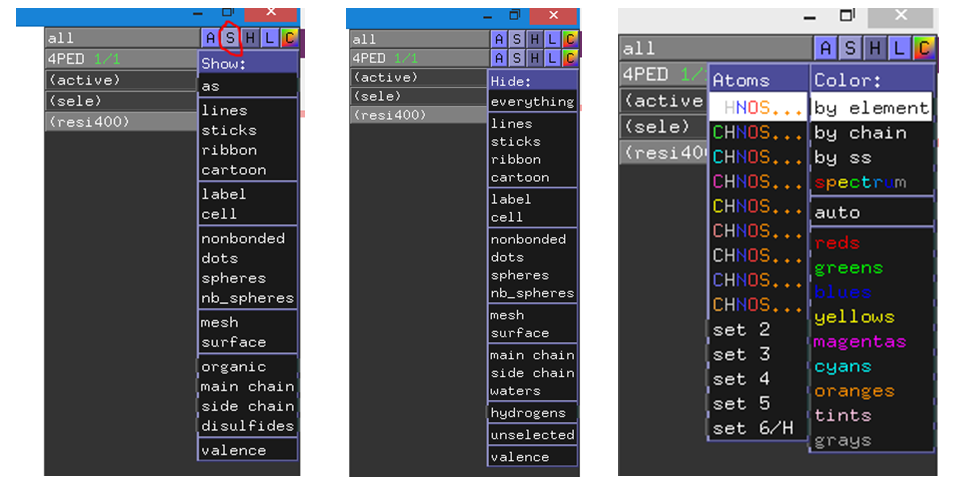
命令行操作
主要包括选择命令、显示/隐藏命令、颜色命令、标签命令、设置命令、图像输出设置命令等。
select命令
select 变量名,选择的原子 #变量名可以是任意的字母或数字组合,select命令可简写为sele;
如何选择原子呢?属性选择符有segi(segment),chain(链),resn(残基名),resi(残基号),name(原子名),symbol(元素符号)。逻辑运算符有 and 或者 & (和)、or (或)、not(非)。
1 |
select ligand,segi lig ##选择segment为lig的所有原子,赋给变量ligand; |
另外还有稍复杂点的运算符 around,expand,within x of ,byres ,neighbor;
1 |
select coor,name fe around 4 #选择铁原子周围4埃内的所有原子(不包括铁原子);s1 around x; |
show命令:显示一种或多种模式
show 显示形式,变量 #也可以不用提前创建变量,直接在这里选择原子`
常用显示形式有lines,sticks,cartoon,ribbon,sphere,surface,mesh
1 |
show sticks,Y24 #显示变量为Y24的所有原子为棍棒形; |
hide命令:操作同show
由于PyMol没有撤销命令,显示命令操作后无法撤销,若之前操作将Y24显示为棍形,现在又不想显示它,可以输入命令hide sticks,Y24
color命令:着色
color 颜色,变量
1 |
color green #整个蛋白着色为绿色; |
label命令
label [selection,[expression]] #selection为选择的原子或对应的变量名,expression为显示的标签文字;
1 |
label resi 24 and name OH,"T24" #在24号残基的OH原子附近标记为T24; |
标签设置
1 |
set label_color,color_name,selection #设置标签颜色 |
添加或删除键
1 |
bond atom1,atom2 #可提前定义变量atom1和atom2,也可以在这里直接输入选择; |
虚键设置
1 |
set dash_length,0.015 #每截虚线的长度 |
叠合命令Align
align source,target #source对象会被移动旋转来叠合到target对象上;
1 |
Align mol1 & resi N1, mol2 & resi N2 #按某一个残基叠合; |
对整个蛋白叠合:鼠标操作:A>>align>>to molecule>>mol2
保存之图片输出
1 |
set ray_shadows,0 #渲染时不显示投影; |
动画视频制作
1 |
load xxx.top,mov,format=top ##pymol要求载入轨迹前要先载入拓扑文件,除非后缀是top,后缀是prmtop的话除非定义一下格式,否则是不认的 |
命令集成
有时我们需要对同一研究体系不同构象作图像显示,或经常打开某一pdb文件,每次输入命令总是太麻烦,为节省时间,可以将所有要操作的命令写到文本文件name.pml中,然后在PyMol命令框中输入@name.pml,就可以执行文本中的命令。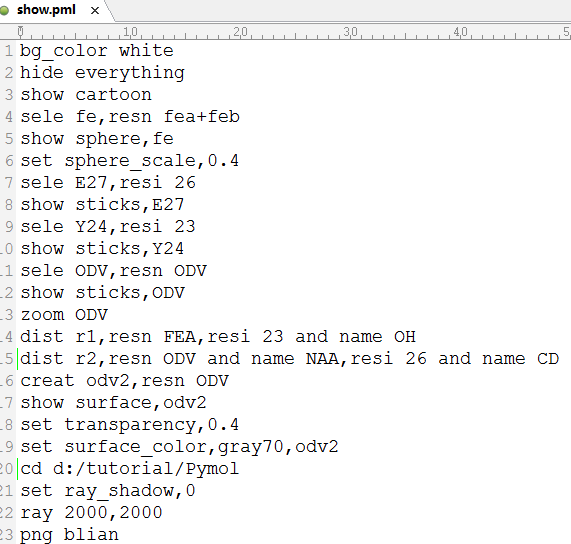
常用命令:
load *.pdb
remove solvent
select str1, resn Cl-
remove str1
align WT_ligs, WT_box_wat_eq3 #后面的是target
alignto WT_ligs #把所有pdb都align到WT_ligs
show sticks, resi 87
set label_size, -1.4
set label_color, black, selection
select selection, resid 84+85+86+94+98+101+151+273+274+278+282+285+336+341+342+345+346+348+418+421+422+423+425+426+430
png image.png, 10cm, dpi=300, ray=1
png image.png, width=3000, height=2400, dpi=300, ray=1
高精度做图命令:
hide
hide labels, agi
show sticks, lig | res #棒状显示lig和res对象,"|"为逻辑“或”
select 5A, byres ligand around 5 #选取ligand5A范围内残基
hide sticks, h. #隐藏H原子
zoom lig | res #居中放大lig和res,其他对象虚化
color wheat, lig & name C* #将lig的碳原子(以C开头的原子名)设为小麦色
set label_size, 30
set label_font_id, 9
#########
import center_of_mass
select AGI1, resn AGI & name C2+C3+C4+C5+C6
com AGI1, object=p1
select AGI2, resn AGI & name C7+C8+C9+C10+C11
com AGI2, object=p2
hide everything, p*
show spheres, p*
set sphere_scale, 0.4
hide labels, measure*
set dash_length, 0.3
set dash_radius, 0.08
set dash_gap, 0.3
color green, measure01
color gray70, measure02
bg_color white
show cartoon
#########
color smudge, lig & name C*
set cartoon_transparency, 0.5
set cartoon_round_helices, 1
set cartoon_fancy_helices, 1
set cartoon_fancy_sheets, 1
set cartoon_color, green, FLS_TPP_DHA & chain A #设置不同链的cartoon不同颜色
set stick_ball #显示棍棒模型
set stick_ball_ratio, 1.3
set stick_h_scale, 0.8
unset opaque_background # 设置背景透明,只在ray下有用
#########
save focus.pse
png label.png
hide labels
png focus.png, 16.93cm, 16.93cm, dpi=300, ray=1
zoom
set cartoon_transparency, 0
png overview.png, 20cm, 18cm, dpi=300, ray=1
save overview.pse
高阶命令:
load candidate.pdb, c_run4
select run4_AGI, c_run4 & resn AGI
sel active_run4, byres c_run4 within 5 of run4_AGI
show sticks, active_run4
show surface, active_run4
show surface, byres c_run4 within 5 of run4_AGI
显示双键:
# In editing mode: select the bond using Ctrl-right-click, then enter:
unbond pk1,pk2
bond pk1,pk2,4
# 1: single bond, 2: double bond, 3:triple bond, 4:delocalized,
...
额外记录一些颜色:
set_color high_lddt_c, [0,0.325490196078431,0.843137254901961 ]
set_color normal_lddt_c, [0.341176470588235,0.792156862745098,0.976470588235294]
set_color medium_lddt_c, [1,0.858823529411765,0.070588235294118]
set_color low_lddt_c, [1,0.494117647058824,0.270588235294118]
