当前位置:
X-MOL 学术
›
Microchem. J.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Fabricating periodic sandwich SERS structure in detecting SARS-CoV-2 S protein with high-performance
Microchemical Journal ( IF 4.9 ) Pub Date : 2024-04-23 , DOI: 10.1016/j.microc.2024.110619 Yansheng Liu , Haonan Hu , Xiaobo Jia , Jin Zhou , Hongli Li , Xiaohong Wang , Shaohui Zhang , Haixin Chang , Fuchong Li , Guofu Wang
Microchemical Journal ( IF 4.9 ) Pub Date : 2024-04-23 , DOI: 10.1016/j.microc.2024.110619 Yansheng Liu , Haonan Hu , Xiaobo Jia , Jin Zhou , Hongli Li , Xiaohong Wang , Shaohui Zhang , Haixin Chang , Fuchong Li , Guofu Wang
|
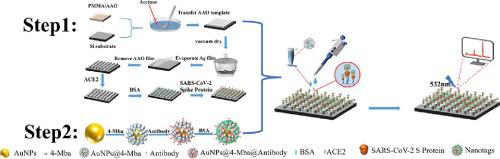
This study reported a sandwich SERS structure in detecting SARS-CoV-2 spike protein (SARS-CoV-2 S protein) with high performance. The periodic Ag nanoarrays (AgNAs) functionalized with angiotensin-converting enzyme 2 (ACE2) were utilized as the bottom structure to specifically bound to the SARS-CoV-2 S protein. A complex of SARS-CoV-2 S protein antibody and 4-mercaptobenzoic acid (4-Mba) modified Au nanoparticles (AuNPs@4-Mba@Antibody) were used as nanotags and top SERS enhancing structures. By utilized the “hot spots” in parallel and perpendicular directions, the SERS signals of the 4-Mba as reporters were largely enhanced. By modulated the diameters and the heights of the AgNAs, the SERS performance of the sandwich structures was optimized by using R6G as a stand analyst with limits of detection (LOD) of 1 × 10−9 M. By applied the optimized sandwich structures in detecting SARS-CoV-2 S protein in phosphate buffered saline (PBS) and pharyngeal swabs solution (PSS), the proposed sandwich structure illustrates a high performance, and the LODs were 0.1 fg mL−1 and 1 fg mL−1 , respectively. This study presented an efficient way to construct a sandwich SERS structure in directly detecting SARS-CoV-2 S protein and this method illustrates a good potential in viral detection.
中文翻译:

构建周期性夹心 SERS 结构以高性能检测 SARS-CoV-2 S 蛋白
本研究报道了一种夹心 SERS 结构在检测 SARS-CoV-2 刺突蛋白 (SARS-CoV-2 S 蛋白) 方面表现出色。用血管紧张素转换酶 2 (ACE2) 功能化的周期性 Ag 纳米阵列 (AgNAs) 被用作与 SARS-CoV-2 S 蛋白特异性结合的底部结构。SARS-CoV-2 S 蛋白抗体和 4-巯基苯甲酸 (4-Mba) 修饰的 Au 纳米颗粒 (AuNPs@4-Mba@Antibody) 的复合物被用作纳米标签和顶部 SERS 增强结构。通过利用平行和垂直方向的“热点”,4-Mba 作为记者的 SERS 信号在很大程度上得到了增强。通过调节 AgNAs 的直径和高度,使用 R6G 作为检测限 (LOD) 为 1 × 10−9 M 的林分分析员,优化了夹层结构的 SERS 性能。通过应用优化的夹心结构检测磷酸盐缓冲盐水 (PBS) 和咽拭子溶液 (PSS) 中的 SARS-CoV-2 S 蛋白,所提出的夹心结构表现出高性能,LOD 分别为 0.1 fg mL-1 和 1 fg mL-1。本研究提出了一种构建夹心 SERS 结构直接检测 SARS-CoV-2 S 蛋白的有效方法,该方法在病毒检测中具有良好的潜力。
更新日期:2024-04-23
中文翻译:

构建周期性夹心 SERS 结构以高性能检测 SARS-CoV-2 S 蛋白
本研究报道了一种夹心 SERS 结构在检测 SARS-CoV-2 刺突蛋白 (SARS-CoV-2 S 蛋白) 方面表现出色。用血管紧张素转换酶 2 (ACE2) 功能化的周期性 Ag 纳米阵列 (AgNAs) 被用作与 SARS-CoV-2 S 蛋白特异性结合的底部结构。SARS-CoV-2 S 蛋白抗体和 4-巯基苯甲酸 (4-Mba) 修饰的 Au 纳米颗粒 (AuNPs@4-Mba@Antibody) 的复合物被用作纳米标签和顶部 SERS 增强结构。通过利用平行和垂直方向的“热点”,4-Mba 作为记者的 SERS 信号在很大程度上得到了增强。通过调节 AgNAs 的直径和高度,使用 R6G 作为检测限 (LOD) 为 1 × 10−9 M 的林分分析员,优化了夹层结构的 SERS 性能。通过应用优化的夹心结构检测磷酸盐缓冲盐水 (PBS) 和咽拭子溶液 (PSS) 中的 SARS-CoV-2 S 蛋白,所提出的夹心结构表现出高性能,LOD 分别为 0.1 fg mL-1 和 1 fg mL-1。本研究提出了一种构建夹心 SERS 结构直接检测 SARS-CoV-2 S 蛋白的有效方法,该方法在病毒检测中具有良好的潜力。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号