序列及长度定制化环状单链DNA的制备及大尺寸DNA折纸结构的构建
DNA作为一种天然的生物大分子以其独特的化学结构和特有的分子间相互作用,在构建功能性纳米结构方面具有得天独厚的优势。利用碱基互补配对作用,DNA分子可以自组装形成具有纳米级精度的有序结构。这种DNA纳米结构还可以在特定的位置对DNA链进行修饰,使之作为支架引导其它分子或纳米材料进行可控自组装。1
2006年加州理工学院的Rothemund提出了DNA折纸术这一全新的DNA自组装策略,将DNA自组装领域推进到一个新的发展阶段。2 该方法使用一条长单链DNA作为骨架链,上百条序列不同的短链DNA作为订书钉链。通过碱基互补作用,订书钉链将骨架链折叠成为所设计的各种形状。在DNA折纸术中,所有订书钉链的DNA序列不同,从而使整个纳米结构在空间上具有完全的可寻址性。与传统的DNA自组装方式相比,DNA折纸术不仅能够在纳米尺度上进行更为精确的组装与排列,得到更加复杂精细的可编程图案与结构,而且实验操作更为简便、组装效率更高。DNA折纸术组装得到的结构可以作为模板,引导其他纳米材料、药物小分子、生物大分子等成分的排列,从而获得光学、电磁学等性能可控的纳米元件、药物载体以及纳米机器人等,使DNA折纸术在纳米领域具有非常广泛的应用价值。3-7
然而,由于天然存在的DNA长单链种类稀少,DNA折纸结构绝大多数使用M13噬菌体的基因组DNA作为骨架链,其长度仅为7000多个碱基,造成DNA折纸结构尺寸受限:利用M13 DNA作为骨架链构筑的正方形结构尺寸仅约为90 nm×90 nm。此外,由于几乎所有的DNA折纸结构均使用同一骨架链,多个折纸结构同时自组装时将存在相互干扰,因而无法在同一反应体系中同时进行多个不同折纸结构的自组装。针对DNA折纸术骨架链尺寸和序列种类受限的问题,研究者开始尝试制备更长的单链DNA作为DNA折纸术的骨架链,从而获得更大尺寸的折纸结构。

序列及长度定制化环状单链DNA的制备及其用于构建大尺寸DNA折纸结构的示意图
近日,南京大学现代工程与应用科学学院的李喆教授(点击查看介绍)与于涵洋教授(点击查看介绍)合作,在ACS Applied Materials & Interfaces 上发表文章,报道了一种序列及长度定制化环状单链DNA制备的方法及其在构建大尺寸DNA折纸结构中的应用。他们首先通过Gibson assembly无缝克隆的方式,将不同来源的特定DNA片段与噬菌体的复制起始位点M13 origin以及质粒的复制起点pBR322 origin进行拼接,组装成双链环状的重组噬菌粒,然后将此重组噬菌粒转化引入包含F因子的大肠杆菌,通过大肠杆菌的繁殖进行大量扩增。经辅助噬菌体侵染后,重组噬菌粒以单链的形式包裹于噬菌体并被分泌到培养液中,经历一系列收集纯化操作后,便可获得相应序列的环状单链DNA。通过该方法,他们制备了4种不同长度的单链DNA序列,包括长度分别为30000多和20000多及另外两种不同序列的、长度为10000多个碱基的单链环状DNA。最后,他们以这4种单链DNA作为骨架链分别成功构建了几种不同的大尺寸DNA矩形和三角形折纸结构。
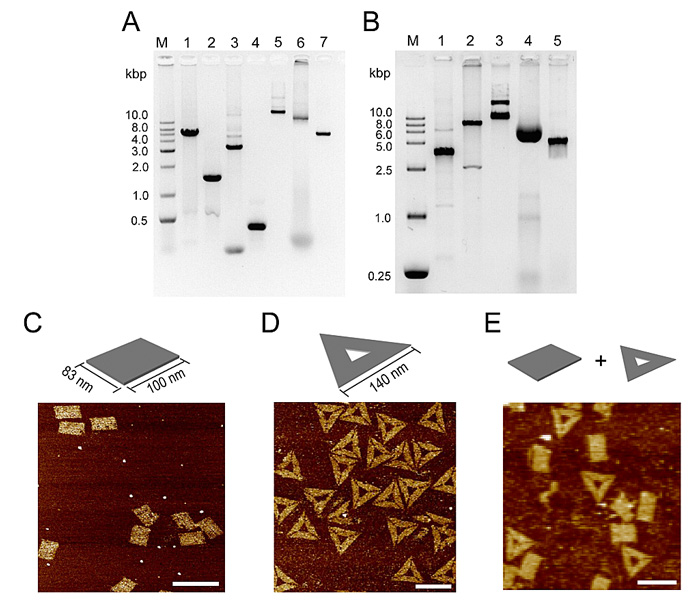
两种含不同序列、长度均为10000多个碱基的单链DNA的制备以及在同一反应体系中同时组装形成两种不同的纳米结构
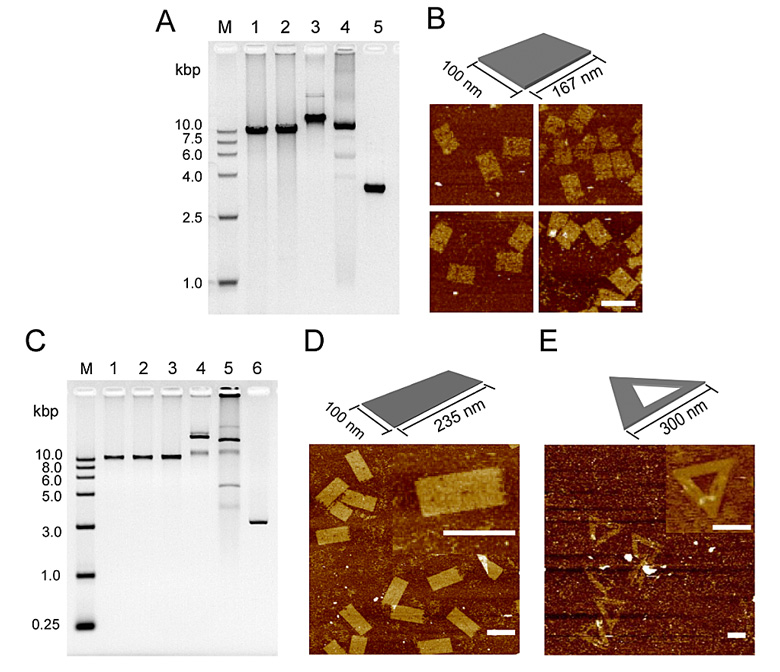
长度为20000多和30000多个碱基单链DNA的制备以及用作骨架链构筑更大尺寸的矩形和三角形折纸结构
该研究提出了一种新型的环状单链DNA的制备方法,该方法不仅可以突破传统的基于M13噬菌体基因组DNA折纸结构的尺寸局限性,而且可以极大地丰富DNA折纸术骨架链的来源。该方法还可以实现在同一反应体系中同时构建多种不同的DNA折纸结构,为构建更为复杂、更大规模的DNA折纸结构提供新的途径。
南京大学现代工程与应用科学学院的博士生陈小星和汪倩为论文的共同第一作者,李喆教授和于涵洋教授为论文的共同通讯作者。该研究工作得到国家自然科学基金青年基金(21603100,21708018)与国家重点研发计划(2016YFA0502600)等项目的资助。
该论文作者为:Xiaoxing Chen, Qian Wang, Jin Peng, Qipeng Long, Hanyang Yu and Zhe Li
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Self-Assembly of Large DNA Origami with Custom-Designed Scaffolds
ACS Appl. Mater. Interfaces, 2018, 10, 24344, DOI: 10.1021/acsami.8b09222
导师介绍
李喆
http://www.x-mol.com/university/faculty/22062
于涵洋
http://www.x-mol.com/university/faculty/22066
参考文献:
1. Liu, X.; Zhang, F.; Jing, X., et al., Complex silica composite nanomaterials templated with DNA origami. Nature, 2018, 559, 593-598.
2. Rothemund, P. W., Folding DNA to create nanoscale shapes and patterns. Nature, 2006, 440, 297-302.
3. Lee, H.; Lytton-Jean, A. K.; Chen, Y., et al., Molecularly self-assembled nucleic acid nanoparticles for targeted in vivo siRNA delivery. Nat. Nanotechnol., 2012, 7, 389-393.
4. Xu, W.; Nathwani, B.; Lin, C., et al., A Programmable DNA Origami Platform to Organize SNAREs for Membrane Fusion. J. Am. Chem. Soc., 2016, 138, 4439-4447.
5. Zhang, H.; Chao, J.; Pan, D., et al., DNA origami-based shape IDs for single-molecule nanomechanical genotyping. Nat. Commun., 2017, 8, 14738.
6. Kuzyk, A.; Schreiber, R.; Fan, Z., et al., DNA-based self-assembly of chiral plasmonic nanostructures with tailored optical response. Nature, 2012, 483, 311-314.
7. Sun, W.; Boulais, E.; Hakobyan, Y., et al., Casting inorganic structures with DNA molds. Science, 2014, 346, 1177-1183.
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!



















































 京公网安备 11010802027423号
京公网安备 11010802027423号