湖南大学聂舟团队Angew:工程化Anti-CRISPR蛋白创建CRISPR-Cas蛋白开关用于激活型基因编辑和病毒蛋白酶检测
具有可编程响应功能的蛋白开关在生物过程监测与控制中发挥着重要作用。CRISPR-Cas系统因其RNA引导Cas蛋白对靶标核酸的可编程结合与处理能力,广泛应用于基因编辑、生物成像和分子诊断。然而,目前缺乏对Cas蛋白功能的有效控制,特别是脱靶活性的风险,这凸显了开发可控CRISPR-Cas系统的必要性。在自然界中,噬菌体与细菌的进化竞争孕育了抗CRISPR(Acr)蛋白,用于抑制Cas蛋白活性以避免CRISPR-Cas免疫系统的攻击。这些天然Acr蛋白为直接控制Cas蛋白功能提供了工具,但其在构建响应型Cas蛋白中的应用受到固有缺陷(如易于二聚化、高结合亲和力、酶催化不可逆抑制等)的限制。
湖南大学聂舟(点击查看介绍)课题组利用计算化学指导的理性蛋白质工程,以抗CRISPR(Acr)蛋白为模块,设计与构建Cas蛋白开关(CasPSs)。通过分子指纹分析、静电势映射和结合自由能计算,精准改造Cas12a与AcrVA4、AcrVA5间的相互作用界面,获得正交的蛋白酶响应型CasPSs。这些CasPSs可将特定蛋白水解事件转化为Cas12a功能激活,实现了哺乳动物细胞中蛋白水解诱导的基因组编辑及病毒感染中病毒蛋白酶活性的灵敏检测。该研究为开发用于可控基因操纵与临床诊断的CRISPR-Cas工具提供了新策略。近日,该成果发表在Angew. Chem. Int. Ed.上,文章第一作者为课题组已毕业博士研究生康文渊。

Scheme 1. 计算化学指导的CasPS理性设计示意图。以AcrVA4为模型,基于对Cas12a和Acr蛋白分子相互作用界面的综合分析与调控,融合这两个模块构建了CasPS,实现了可激活的基因组编辑和病毒蛋白酶检测。
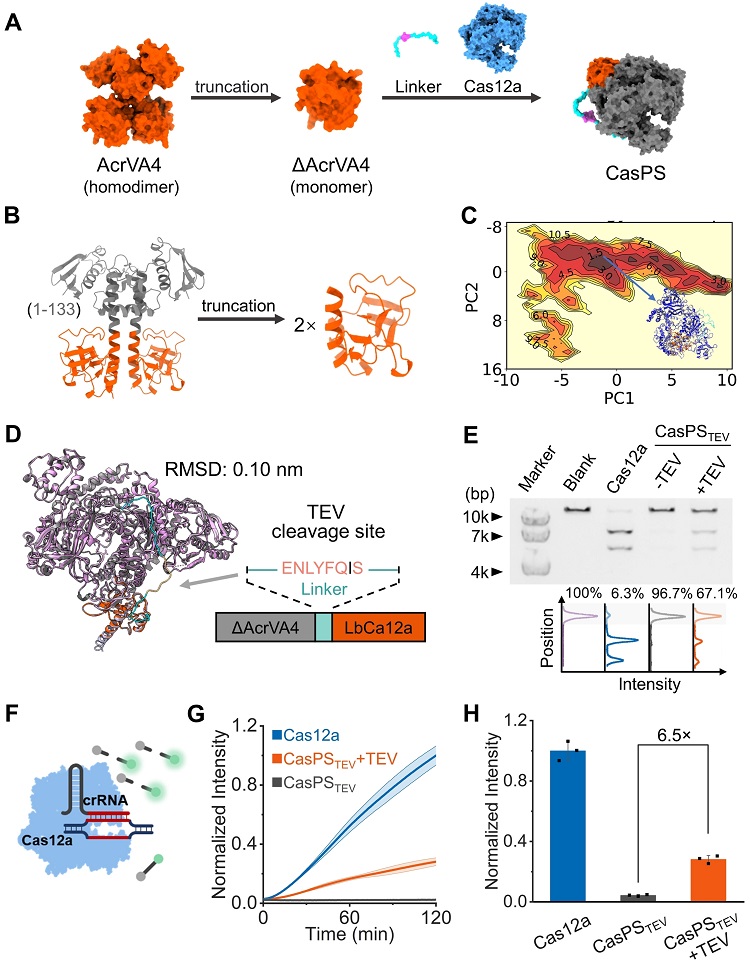
图1. 融合AcrVA4、TEV蛋白酶识别序列与Cas12a构建CasPS并探究其基本性质。基于Cas12a与AcrVA4的相互作用,构建了模仿天然酶原激活机制的Cas蛋白开关(CasPS)。通过理性融合Cas蛋白与Acr蛋白,实现了蛋白酶响应型CasPS的构建(图1A)。针对AcrVA4的二聚化倾向可能干扰CasPS激活的问题,截短了AcrVA4(1-133位),这既保留了其抑制活性又避免了二聚现象(图1B)。以Cas12a/AcrVA4配合物的晶体结构作为参考,分析了空间取向并确定了融合方式。通过分子动力学模拟,验证了融合操作对蛋白稳定性的影响,确保融合后的CasPS稳定性不变(图1C和D)。进一步表达纯化了CasPSTEV,并对其开关功能进行了评估。实验结果显示,CasPSTEV能有效抑制Cas12a的活性,而在TEV蛋白酶的作用下,通过特异性切割识别序列,实现了Cas12a活性由关到开的转换,开关比率为6.5(图1G和H)。
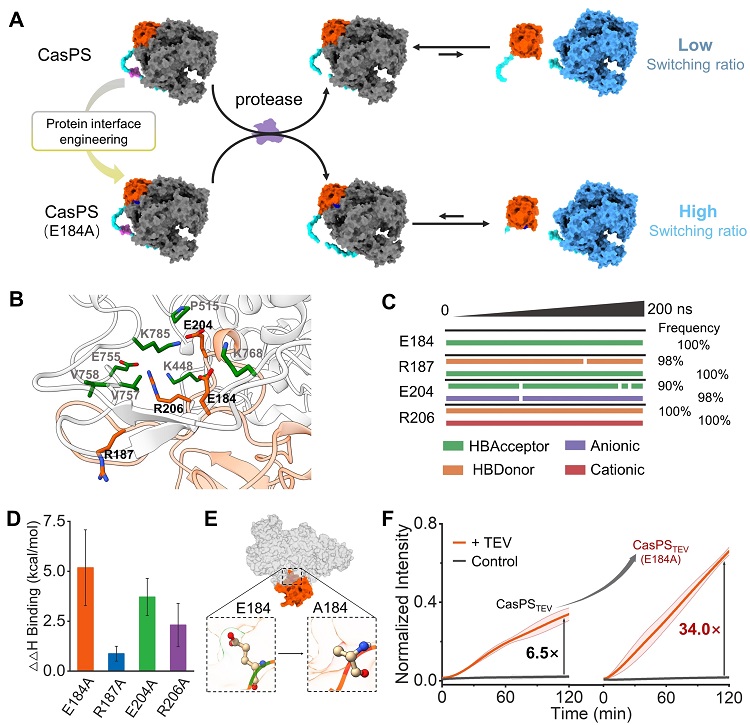
图2. 计算化学指导的蛋白质工程提高CasPS的开关比率。为提升CasPS的开关比率,在计算化学指导下微调了ΔAcrVA4与Cas12a的分子相互作用界面,以精细调控其亲和力(图2A)。基于已知的晶体结构(PDB ID:6OMV),利用分子动力学模拟深入探究了两者间的相互作用,并细致分析了模拟轨迹的分子指纹。分析结果显示,ΔAcrVA4中的E184、R187、E204和R206在与Cas12a的相互作用中呈现出高频接触(≥90%),对亲和力具有显著影响(图2B和C)。进一步计算显示,E184A、R187A、E204A和R206A的突变分别引起了5.18±1.88、0.87±0.37、3.71±0.92和2.31±1.07 kcal/mol的相对结合自由能变化(图2D)。基于上述计算结果,选择变化最为显著的E184进行点突变。实验结果表明,相较于突变前的CasPS,构建的CasPSTEV(E184A)在保持相当背景信号的同时,显著增强了响应信号,从而将开关比率提升至34.0(图2E和F)。

图3. 连接肽长度对CasPS的开关比率影响以及正交响应的CasPSs。我们还探讨了连接肽对CasPS性能的影响(图3A),并测定了连接肽长度在20至40 aa范围内时CasPS的开关比率。尽管所有CasPS变体均显示显著的响应信号,但20 aa的连接肽显著增强了背景信号。AlphaFold2模型分析揭示,短连接肽阻碍了ΔAcrVA4与Cas12a的有效相互作用,削弱了其抑制效果。实验测定的开关比率分别为3.2、34.0和21.6(图3B)。因此,本研究选择30 aa作为优化连接肽长度,以推进后续实验。为验证策略的通用性,构建了基于丙型肝炎病毒(HCV)NS3/4A蛋白酶和人鼻病毒(HRV)14型3C蛋白酶底物序列的CasPSNS3/4A和CasPS3C。经蛋白酶处理后,这些CasPS展现出高开关比率及对相应蛋白酶的特异性响应(图3C-E),证实了该策略的广泛适用性。
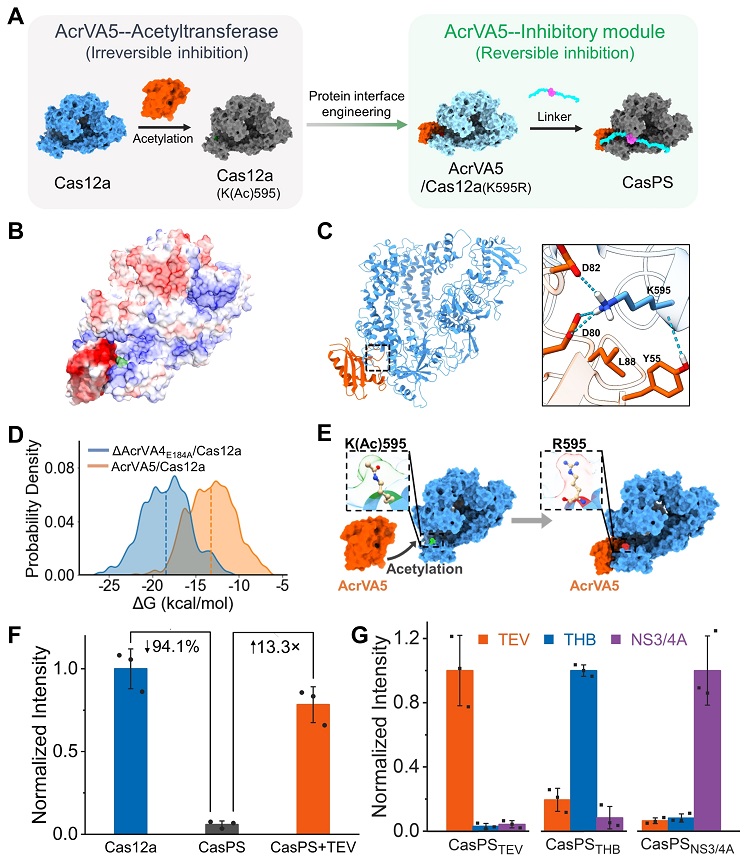
图4. 基于Cas12a与AcrVA5的CasPS及其性质研究。进一步探索了其他Acr蛋白在构建Cas12a开关中的适用性。其中,AcrVA5通过乙酰化Cas12a的PAM结合位点,抑制其与DNA的结合活性,导致功能失活(图4A)。静电势能分析显示AcrVA5催化界面富含负电荷,而Cas12a的PAM结合区域则呈现显著正电荷,暗示两者间可能存在静电相互作用(图4B)。结合蛋白-蛋白对接和静电势能分析,构建了AcrVA5与Cas12a的相互作用模型,发现AcrVA5的疏水残基Y55和L88插入Cas12a内部,形成非极性联系,同时Y55、D80和D82与Cas12a存在氢键和静电作用(图4C)。结合自由能计算表明,AcrVA5/Cas12a复合物的亲和力略弱于AcrVA4(E184A)/Cas12a复合物,揭示了AcrVA5用于构建CasPS的潜力(图4D)。为了消除AcrVA5的不可逆抑制功能,突变Cas12a乙酰化位点(K595R),构建了基于AcrVA5的CasPS(图4E)。与基于AcrVA4的CasPS相比,基于AcrVA5的CasPSTEV的开关比率略低(13.3),但Cas12a活性恢复率较高(78.3%,图4F)。此外,通过替换底物识别位点,验证了开关良好的可拓展性与响应的特异性(图4G)。
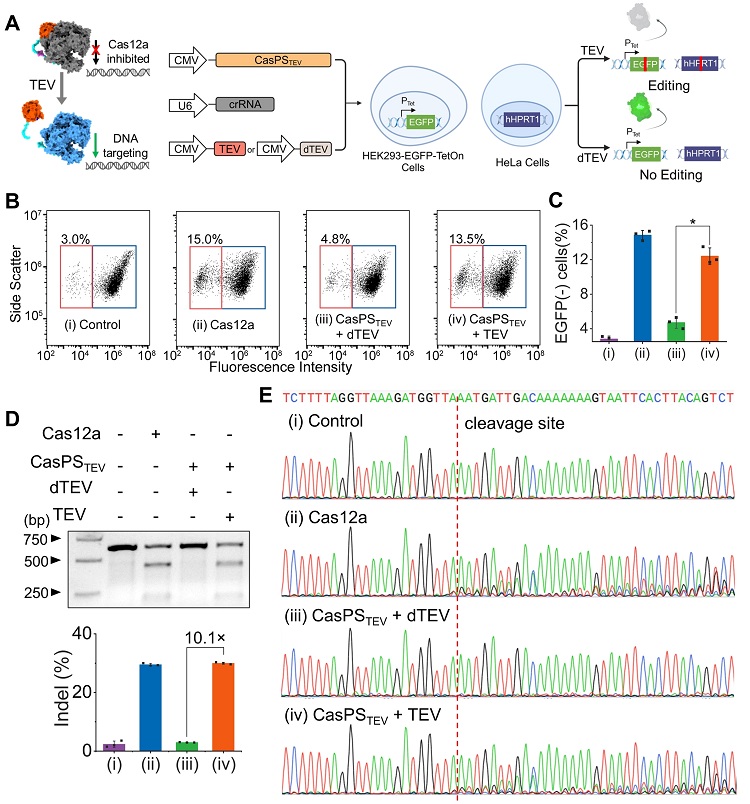
图5. CasPS用于蛋白酶激活的基因编辑。在体外验证特异性蛋白酶水解激活CasPS的有效性后,进一步探讨了其在哺乳动物细胞内的应用潜力。首先在HEK293-EGFP-TetOn细胞系中通过EGFP基因切割实验评估了CasPS在蛋白酶介导的基因组编辑中的应用潜力。实验结果显示,在缺乏蛋白酶激活时,EGFP基因只有微小的沉默效率;而在蛋白酶存在条件下,EGFP基因沉默效率效率与野生型Cas12a相近(图5A-C)。此外,进一步评估了CasPS在HeLa细胞系中对内源性HPRT1基因的编辑能力,采用标准的T7E1检测方法进行定量。结果表明,在蛋白酶激活条件下,CasPS的编辑效率与Cas12a相当,并展现出约10.1倍的开关比率(图5D和E)。综上所述,实验表明CasPS可实现特定蛋白酶触发的可激活基因编辑,为精准基因工程提供了一种新的策略。

图6. 基于CasPS的蛋白酶分析新方法及其在生物样品中病毒蛋白酶高灵敏检测的应用。CRISPR-Cas12a的反式切割活性在核酸分子诊断中显示出巨大潜力,但其在蛋白酶标志物检测中的应用尚未深入探索。鉴于病毒蛋白酶在病毒感染中的核心作用,系统研究了CasPS在病毒感染检测中的应用。通过病毒蛋白酶介导的CasPS切割,成功恢复了Cas12a的活性,并利用其持续切割单链DNA报告基因实现信号的高效放大,进而通过荧光或试纸条实现信号的快速读取(图6A)。实验结果显示,基于CasPS的3C蛋白酶检测限低于1×10-3 U/μL,相较于传统多肽探针方法,检测灵敏度提升了20倍以上,且信号强度显著增强,达到9.6-28倍(图6B-C)。此外,还通过市售抑制剂验证了CasPS在检测3C蛋白酶抑制方面的实用性(图6D)。我们进一步将CasPS系统成功应用于柯萨奇病毒B3(CVB3)感染细胞的检测,通过荧光和试纸条信号有效区分感染与未感染细胞。荧光信号与病毒载量和感染时间呈正相关(图6 F-J),在40个生物样本测试中,AUC值高达0.942,显示出优秀的敏感性与特异性(图6 K和L)。总之,这些数据有力证明了基于CasPS的检测方法在复杂生物样品中检测病毒感染的可行性和显著优势。
总结与展望
综上所述,聂舟课题组借鉴酶原自抑制与激活机制,利用Acr蛋白作为模块化组件,成功开发出可编程的Cas蛋白开关。通过计算化学指导的蛋白质界面工程,克服了Acr在构建蛋白开关时面临的挑战,创建了具有可编程响应功能的正交CasPSs,无需繁琐的筛选过程。该方法实现了Cas12a蛋白开关的便捷模块化生成,具备可编程和可定制的特性。实验证明,这些CasPSs在哺乳动物细胞中展示了高效的可激活基因组编辑能力,并能对病毒感染期间的靶标蛋白酶活性进行灵敏分析。本研究策略基于Cas12a与Acr蛋白之间的分子界面相互作用,通过微调Cas-Acr蛋白对,有望扩展至其他第二类Cas蛋白。因此,本研究提出了一种基于计算化学指导的理性构建Cas蛋白开关的方法,为基因编辑、生物化学调控及临床诊断领域中的可控CRISPR-Cas工具开发提供了高效的策略。
原文(扫描或长按二维码,识别后直达原文页面,或点此查看原文):

Engineering Anti-CRISPR Proteins to Create CRISPR-Cas Protein Switches for Activatable Genome Editing and Viral Protease Detection
Wenyuan Kang, Fei Xiao, Xi Zhu, Xinyu Ling, Shiyi Xie, Ruimiao Li, Peihang Yu, Linxin Cao, Chunyang Lei, Ye Qiu, Tao Liu, Zhou Nie
Angew. Chem. Int. Ed., 2024, DOI: 10.1002/anie.202400599
导师介绍
聂舟
https://www.x-mol.com/university/faculty/10090
如果篇首注明了授权来源,任何转载需获得来源方的许可!如果篇首未特别注明出处,本文版权属于 X-MOL ( x-mol.com ), 未经许可,谢绝转载!






























 京公网安备 11010802027423号
京公网安备 11010802027423号